Enzyme
Les enzymes ( / ˈ ɛ n z aɪ m z / ) sont des protéines qui agissent comme des catalyseurs biologiques (biocatalyseurs). Les catalyseurs accélèrent les Réactions chimiques . Les molécules sur lesquelles les enzymes peuvent agir sont appelées substrats , et l’enzyme convertit les substrats en différentes molécules appelées produits . Presque tous les processus métaboliques dans la cellule ont besoin d’ une catalyse enzymatique pour se produire à des taux suffisamment rapides pour maintenir la vie. [1] : 8.1 Voies métaboliques dépendent des enzymes pour catalyser les étapes individuelles. L’étude des enzymes s’appelle l’ enzymologie et le domaine de l’analyse des pseudoenzymes reconnaît qu’au cours de l’évolution, certaines enzymes ont perdu la capacité d’effectuer une catalyse Biologique, ce qui se reflète souvent dans leurs séquences d’acides aminés et leurs propriétés « pseudocatalytiques » inhabituelles. [2] [3]
 L’enzyme Glucosidase convertit le sucre maltose en deux sucres de glucose . Résidus du site actif en rouge, substrat de maltose en noir et cofacteur NAD en jaune. ( APB : 1OBB )
L’enzyme Glucosidase convertit le sucre maltose en deux sucres de glucose . Résidus du site actif en rouge, substrat de maltose en noir et cofacteur NAD en jaune. ( APB : 1OBB )
Les enzymes sont connues pour catalyser plus de 5 000 types de réactions biochimiques. [4] D’autres biocatalyseurs sont des molécules d’ARN catalytiques , appelées ribozymes. La spécificité des enzymes vient de leurs structures tridimensionnelles uniques .
Comme tous les catalyseurs, les enzymes augmentent la Vitesse de réaction en diminuant son énergie d’activation . Certaines enzymes peuvent faire en sorte que leur conversion du substrat en produit se produise plusieurs millions de fois plus rapidement. Un exemple extrême est l’ orotidine 5′-phosphate décarboxylase , qui permet à une réaction qui autrement prendrait des millions d’années de se produire en quelques millisecondes. [5] [6] Chimiquement, les enzymes sont comme n’importe quel Catalyseur et ne sont pas consommées dans les Réactions chimiques, ni ne modifient l’ équilibre d’une réaction. Les enzymes diffèrent de la plupart des autres catalyseurs en étant beaucoup plus spécifiques. L’activité enzymatique peut être affectée par d’autres molécules : les inhibiteurs sont des molécules qui diminuent l’activité enzymatique, etles activateurs sont des molécules qui augmentent l’activité. De nombreux médicaments et poisons thérapeutiques sont des inhibiteurs d’enzymes. L’activité d’une enzyme diminue nettement en dehors de sa température et de son pH optimaux , et de nombreuses enzymes sont (en permanence) dénaturées lorsqu’elles sont exposées à une chaleur excessive, perdant leur structure et leurs propriétés catalytiques.
Certaines enzymes sont utilisées commercialement, par exemple dans la synthèse d’ Antibiotiques . Certains produits ménagers utilisent des enzymes pour accélérer les Réactions chimiques : les enzymes des lessives biologiques décomposent les taches de protéines, d’amidon ou de graisse sur les vêtements, et les enzymes des attendrisseurs à viande décomposent les protéines en molécules plus petites, ce qui rend la viande plus facile à mâcher.
Étymologie et histoire

 Edouard Büchner
Edouard Büchner
À la fin du XVIIe et au début du XVIIIe siècle, la digestion de la viande par les sécrétions gastriques [7] et la conversion de l’amidon en sucres par les extraits de plantes et la salive étaient connues, mais les mécanismes par lesquels elles se produisaient n’avaient pas été identifiés. [8]
Le chimiste français Anselme Payen fut le premier à découvrir une enzyme, la diastase , en 1833. [9] Quelques décennies plus tard, en étudiant la fermentation du sucre en alcool par la levure , Louis Pasteur conclut que cette fermentation était causée par une force vitale contenue dans les cellules de levure appelées “ferments”, dont on pensait qu’elles ne fonctionnaient qu’au sein d’organismes vivants. Il écrit que « la fermentation alcoolique est un acte corrélé à la vie et à l’organisation des cellules de levure, et non à la mort ou à la putréfaction des cellules ». [dix]
En 1877, le physiologiste allemand Wilhelm Kühne (1837–1900) a utilisé pour la première fois le terme enzyme , qui vient du grec ἔνζυμον , «levé» ou «dans la levure», pour décrire ce processus. [11] Le mot enzyme a été utilisé plus tard pour désigner des substances non vivantes telles que la pepsine , et le mot ferment a été utilisé pour désigner l’activité chimique produite par des organismes vivants. [12]
Eduard Buchner a soumis son premier article sur l’étude des extraits de levure en 1897. Dans une série d’expériences à l’ Université de Berlin , il a découvert que le sucre était fermenté par des extraits de levure même lorsqu’il n’y avait pas de cellules de levure vivantes dans le mélange. [13] Il a nommé l’enzyme qui a provoqué la fermentation du saccharose ” zymase “. [14] En 1907, il a reçu le prix Nobel de chimie pour “sa découverte de la fermentation acellulaire”. Suivant l’exemple de Buchner, les enzymes sont généralement nommées en fonction de la réaction qu’elles réalisent : le suffixe -ase est associé au nom du substrat (par exemple, lactaseest l’enzyme qui clive le lactose ) ou au type de réaction (par exemple, l’ADN polymérase forme des polymères d’ADN). [15]
L’identité biochimique des enzymes était encore inconnue au début des années 1900. De nombreux scientifiques ont observé que l’activité enzymatique était associée aux protéines, mais d’autres (comme le lauréat du prix Nobel Richard Willstätter ) ont soutenu que les protéines n’étaient que des supports pour les véritables enzymes et que les protéines en soi étaient incapables de catalyse. [16] En 1926, James B. Sumner a montré que l’enzyme uréase était une protéine pure et l’a cristallisée ; il a fait de même pour l’enzyme catalase en 1937. La conclusion que les protéines pures peuvent être des enzymes a été définitivement démontrée par John Howard Northrop et Wendell Meredith Stanley, qui a travaillé sur les enzymes digestives pepsine (1930), trypsine et chymotrypsine . Ces trois scientifiques ont reçu le prix Nobel de chimie en 1946. [17]
La découverte que les enzymes pouvaient être cristallisées a finalement permis de résoudre leurs structures par cristallographie aux rayons X. Cela a d’abord été fait pour le lysozyme , une enzyme présente dans les larmes, la salive et les blancs d’œufs qui digère l’enrobage de certaines bactéries ; la structure a été résolue par un groupe dirigé par David Chilton Phillips et publiée en 1965. [18] Cette structure à haute résolution du lysozyme a marqué le début du domaine de la biologie structurale et l’effort pour comprendre comment les enzymes fonctionnent à un niveau atomique de détail . [19]
Classement et nomenclature
Les enzymes peuvent être classées selon deux critères principaux : soit la similarité des séquences d’acides aminés (et donc la relation évolutive), soit l’activité enzymatique.
Activité enzymatique . Le nom d’une enzyme est souvent dérivé de son substrat ou de la réaction chimique qu’elle catalyse, le mot se terminant par -ase . [1] : 8.1.3 Des exemples sont la lactase , l’alcool déshydrogénase et l’ADN polymérase . Différentes enzymes qui catalysent la même réaction chimique sont appelées isozymes . [1] : 10,3
L’ Union Internationale de Biochimie et de Biologie Moléculaire a élaboré une nomenclature des enzymes, les numéros CE (pour “Enzyme Commission”) . Chaque enzyme est décrite par « EC » suivi d’une séquence de quatre chiffres qui représentent la hiérarchie de l’activité enzymatique (du très général au très spécifique). Autrement dit, le premier chiffre classe largement l’enzyme en fonction de son mécanisme tandis que les autres chiffres ajoutent de plus en plus de spécificité. [20]
La classification de niveau supérieur est :
- EC 1, Oxydoréductases : catalysent les réactions d’Oxydation /réduction
- EC 2, Transférases : transfert d’un groupe fonctionnel ( par exemple un groupe méthyle ou phosphate)
- EC 3, Hydrolases : catalysent l’ hydrolyse de diverses liaisons
- EC 4, Lyases : coupent diverses liaisons par des moyens autres que l’hydrolyse et l’Oxydation
- EC 5, Isomérases : catalysent les changements d’ isomérisation au sein d’une seule molécule
- EC 6, Ligases : joindre deux molécules par des liaisons covalentes .
- EC 7, Translocases : catalysent le mouvement des ions ou des molécules à travers les membranes, ou leur séparation au sein des membranes.
Ces sections sont subdivisées par d’autres caractéristiques telles que le substrat, les produits et le Mécanisme chimique . Une enzyme est entièrement spécifiée par quatre désignations numériques. Par exemple, l’ hexokinase (EC 2.7.1.1) est une transférase (EC 2) qui ajoute un groupe phosphate (EC 2.7) à un sucre hexose, une molécule contenant un groupe alcool (EC 2.7.1). [21]
Similitude de séquence . Les catégories EC ne reflètent pas la similarité des séquences. Par exemple, deux ligases de même numéro EC qui catalysent exactement la même réaction peuvent avoir des séquences complètement différentes. Indépendamment de leur fonction, les enzymes, comme toutes les autres protéines, ont été classées par leur similarité de séquence dans de nombreuses familles. Ces familles ont été documentées dans des dizaines de bases de données de protéines et de familles de protéines différentes telles que Pfam . [22]
Structure

![Un graphique montrant que la <a href='/?s=Vitesse+de+réaction'>Vitesse de réaction</a> augmente de façon exponentielle avec la température jusqu’à ce que la dénaturation la fasse diminuer à nouveau.” height=”148″ src=”” data-src=”//upload.wikimedia.org/wikipedia/commons/thumb/d/d5/Q10_graph_c.svg/400px-Q10_graph_c.svg.png” width=”400″> L’activité enzymatique augmente initialement avec la température ( coefficient Q10 ) jusqu’à ce que la structure de l’enzyme se déploie ( dénaturation ), conduisant à une <a href='/?s=Vitesse+de+réaction'>Vitesse de réaction</a> optimale à une température intermédiaire. </p>
<p>Les enzymes sont généralement des protéines globulaires , agissant seules ou dans des complexes plus grands . La séquence des acides aminés spécifie la structure qui à son tour détermine l’activité catalytique de l’enzyme. <sup>[23]</sup> Bien que la structure détermine la fonction, une nouvelle activité enzymatique ne peut pas encore être prédite à partir de la structure seule. <sup>[24]</sup> Les structures enzymatiques se déplient ( dénaturent ) lorsqu’elles sont chauffées ou exposées à des dénaturants chimiques et cette perturbation de la structure entraîne généralement une perte d’activité. <sup>[25]</sup> La dénaturation enzymatique est normalement liée à des températures supérieures au niveau normal d’une espèce ; en conséquence, les enzymes de bactéries vivant dans des environnements volcaniques tels que des sources chaudessont prisés par les utilisateurs industriels pour leur capacité à fonctionner à des températures élevées, permettant aux réactions catalysées par des enzymes d’être opérées à une vitesse très élevée.</p>
<p>Les enzymes sont généralement beaucoup plus grosses que leurs substrats. Les tailles vont de seulement 62 résidus d’acides aminés, pour le monomère de 4-oxalocrotonate tautomérase , <sup>[26]</sup> à plus de 2 500 résidus dans la synthase d’acide gras animal . <sup>[27]</sup> Seule une petite partie de leur structure (environ 2 à 4 acides aminés) est directement impliquée dans la catalyse : le site catalytique. <sup>[28]</sup> Ce site catalytique est situé à côté d’un ou plusieurs sites de liaison où les résidus orientent les substrats. Le site catalytique et le site de liaison composent ensemble le site actif de l’enzyme. La majorité restante de la structure enzymatique sert à maintenir l’orientation précise et la dynamique du site actif. <sup>[29]</sup></p>
<p>Dans certaines enzymes, aucun acide aminé n’est directement impliqué dans la catalyse ; au lieu de cela, l’enzyme contient des sites pour lier et orienter les cofacteurs catalytiques . <sup>[29]</sup> Les structures enzymatiques peuvent également contenir des sites allostériques où la liaison d’une petite molécule provoque un changement conformationnel qui augmente ou diminue l’activité. <sup>[30]</sup></p>
<p>Il existe un petit nombre de catalyseurs biologiques à base d’ ARN appelés ribozymes , qui peuvent à nouveau agir seuls ou en complexe avec des protéines. Le plus commun d’entre eux est le ribosome qui est un complexe de composants protéiques et catalytiques d’ARN. <sup>[1] : 2,2 </sup></p>
<h2> Mécanisme</h2>
<p> <img loading=](http://upload.wikimedia.org/wikipedia/commons/thumb/2/21/Enzyme_structure.svg/400px-Enzyme_structure.svg.png)
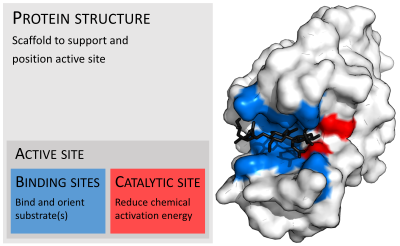 Organisation de la structure enzymatique et exemple de lysozyme . Sites de liaison en bleu, site catalytique en rouge et substrat de peptidoglycane en noir. ( APB : 9LYZ )
Organisation de la structure enzymatique et exemple de lysozyme . Sites de liaison en bleu, site catalytique en rouge et substrat de peptidoglycane en noir. ( APB : 9LYZ )
Liaison au substrat
Les enzymes doivent se lier à leurs substrats avant de pouvoir catalyser une réaction chimique. Les enzymes sont généralement très spécifiques quant aux substrats auxquels elles se lient, puis à la réaction chimique catalysée. La spécificité est obtenue en liant des poches de forme, de charge et de caractéristiques hydrophiles / hydrophobes complémentaires aux substrats. Les enzymes peuvent donc faire la distinction entre des molécules de substrat très similaires pour être chimiosélectives , régiosélectives et stéréospécifiques . [31]
Certaines des enzymes présentant la spécificité et la précision les plus élevées sont impliquées dans la copie et l’ expression du génome . Certaines de ces enzymes ont des mécanismes de ” relecture “. Ici, une enzyme telle que l’ADN polymérase catalyse une réaction dans un premier temps puis vérifie que le produit est correct dans un second temps. [32] Ce processus en deux étapes entraîne des taux d’erreur moyens inférieurs à 1 erreur sur 100 millions de réactions dans les polymérases de mammifères haute fidélité. [1] : 5.3.1 Des mécanismes de relecture similaires sont également trouvés dans l’ARN polymérase , [33] les aminoacyl ARNt synthétases [34] et les ribosomes. [35]
Inversement, certaines enzymes présentent une promiscuité enzymatique , ayant une large spécificité et agissant sur une gamme de différents substrats physiologiquement pertinents. De nombreuses enzymes possèdent de petites activités secondaires qui sont apparues fortuitement (c’est-à-dire de manière neutre ), qui peuvent être le point de départ de la sélection évolutive d’une nouvelle fonction. [36] [37]
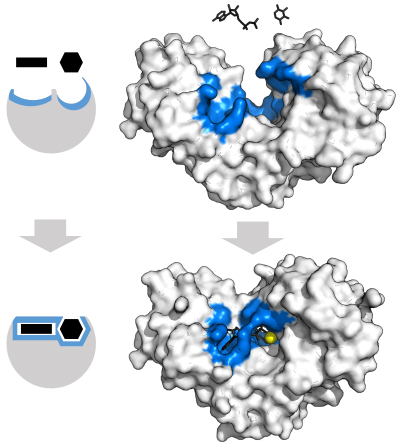
 L’enzyme change de forme par ajustement induit lors de la liaison au substrat pour former un complexe enzyme-substrat. L’hexokinase a un grand mouvement d’ajustement induit qui se referme sur les substrats adénosine triphosphate et xylose . Sites de liaison en bleu, substrats en noir et cofacteur Mg 2+ en jaune. ( APB : 2E2N , 2E2Q ) Modèle “serrure et clé”
L’enzyme change de forme par ajustement induit lors de la liaison au substrat pour former un complexe enzyme-substrat. L’hexokinase a un grand mouvement d’ajustement induit qui se referme sur les substrats adénosine triphosphate et xylose . Sites de liaison en bleu, substrats en noir et cofacteur Mg 2+ en jaune. ( APB : 2E2N , 2E2Q ) Modèle “serrure et clé”
Pour expliquer la spécificité observée des enzymes, Emil Fischer a proposé en 1894 que l’enzyme et le substrat possèdent des formes géométriques complémentaires spécifiques qui s’emboîtent exactement l’une dans l’autre. [38] C’est ce que l’on appelle souvent le modèle “la serrure et la clé”. [1] : 8.3.2 Ce premier modèle explique la spécificité enzymatique, mais ne parvient pas à expliquer la stabilisation de l’état de transition que les enzymes atteignent. [39]
Modèle à ajustement induit
En 1958, Daniel Koshland a suggéré une modification du modèle de la serrure et de la clé : les enzymes étant des structures plutôt flexibles, le site actif est continuellement remodelé par les interactions avec le substrat lorsque le substrat interagit avec l’enzyme. [40] En conséquence, le substrat ne se lie pas simplement à un site actif rigide ; les chaînes latérales d’acides aminés qui composent le site actif sont moulées dans les positions précises qui permettent à l’enzyme d’exercer sa fonction catalytique. Dans certains cas, tels que les glycosidases , la molécule de substrat change également légèrement de forme lorsqu’elle pénètre dans le site actif. [41]Le site actif continue de changer jusqu’à ce que le substrat soit complètement lié, moment auquel la forme finale et la distribution de charge sont déterminées. [42] L’ajustement induit peut améliorer la fidélité de la reconnaissance moléculaire en présence de compétition et de bruit via le mécanisme de relecture conformationnelle . [43]
Catalyse
Les enzymes peuvent accélérer les réactions de plusieurs manières, qui abaissent toutes l’ énergie d’activation (ΔG ‡ , énergie libre de Gibbs ) [44]
- En stabilisant l’état de transition :
- Créer un environnement avec une distribution de charge complémentaire à celle de l’état de transition pour abaisser son énergie [45]
- En fournissant une voie de réaction alternative :
- Réagissant temporairement avec le substrat, formant un intermédiaire covalent pour fournir un état de transition énergétique inférieur [46]
- En déstabilisant l’état fondamental du substrat :
- Déformer le ou les substrats liés dans leur forme d’état de transition pour réduire l’énergie nécessaire pour atteindre l’état de transition [47]
- En orientant les substrats dans un arrangement productif pour réduire le changement d’ entropie de la réaction [48] (la contribution de ce mécanisme à la catalyse est relativement faible) [49]
Les enzymes peuvent utiliser plusieurs de ces mécanismes simultanément. Par exemple, les protéases telles que la trypsine effectuent une catalyse covalente à l’aide d’une triade catalytique , stabilisent l’accumulation de charge sur les états de transition à l’aide d’un trou oxyanion , complètent l’ hydrolyse à l’aide d’un substrat d’eau orienté. [50]
Dynamique
Les enzymes ne sont pas des structures rigides et statiques ; au lieu de cela, ils ont des mouvements dynamiques internes complexes – c’est-à-dire des mouvements de parties de la structure de l’enzyme telles que des résidus d’acides aminés individuels, des groupes de résidus formant une boucle protéique ou une unité de structure secondaire , voire un domaine protéique entier . Ces mouvements donnent naissance à un ensemble conformationnel de structures légèrement différentes qui s’interconvertissent à l’ équilibre . Différents états au sein de cet ensemble peuvent être associés à différents aspects de la fonction d’une enzyme. Par exemple, différentes conformations de l’enzyme dihydrofolate réductasesont associés aux étapes de liaison au substrat, de catalyse, de libération de cofacteur et de libération de produit du cycle catalytique, [51] conformément à la théorie de la résonance catalytique .
Présentation du substrat
La présentation du substrat est un processus où l’enzyme est séquestrée loin de son substrat. Les enzymes peuvent être séquestrées dans la membrane plasmique loin d’un substrat dans le noyau ou le cytosol. Ou à l’intérieur de la membrane, une enzyme peut être séquestrée dans des radeaux lipidiques loin de son substrat dans la région désordonnée. Lorsque l’enzyme est libérée, elle se mélange à son substrat. Alternativement, l’enzyme peut être séquestrée près de son substrat pour activer l’enzyme. Par exemple, l’enzyme peut être soluble et lors de l’activation se lier à un lipide dans la membrane plasmique et ensuite agir sur des molécules dans la membrane plasmique.
Modulation allostérique
Les sites allostériques sont des poches sur l’enzyme, distinctes du site actif, qui se lient aux molécules de l’environnement cellulaire. Ces molécules provoquent alors une modification de la conformation ou de la dynamique de l’enzyme qui est transduite vers le site actif et affecte ainsi la Vitesse de réaction de l’enzyme. [52] De cette manière, les interactions allostériques peuvent inhiber ou activer des enzymes. Les interactions allostériques avec les métabolites en amont ou en aval de la voie métabolique d’une enzyme provoquent une régulation par rétroaction , modifiant l’activité de l’enzyme en fonction du flux à travers le reste de la voie. [53]
Cofacteurs

 Structure chimique du pyrophosphate de thiamine et structure protéique de la transcétolase . Cofacteur pyrophosphate de thiamine en jaune et substrat xylulose 5-phosphate en noir. ( APB : 4KXV )
Structure chimique du pyrophosphate de thiamine et structure protéique de la transcétolase . Cofacteur pyrophosphate de thiamine en jaune et substrat xylulose 5-phosphate en noir. ( APB : 4KXV )
Certaines enzymes n’ont pas besoin de composants supplémentaires pour montrer leur pleine activité. D’autres nécessitent que des molécules non protéiques appelées cofacteurs soient liées pour l’activité. [54] Les cofacteurs peuvent être soit des composés inorganiques (par exemple, des ions métalliques et des amas fer-soufre ) soit des composés organiques (par exemple, la flavine et l’ hème ). Ces cofacteurs servent à plusieurs fins; par exemple, les ions métalliques peuvent aider à stabiliser les espèces nucléophiles dans le site actif. [55] Les cofacteurs organiques peuvent être soit des coenzymes , qui sont libérées du site actif de l’enzyme pendant la réaction, soit des groupes prosthétiques, qui sont étroitement liés à une enzyme. Les groupes prosthétiques organiques peuvent être liés de manière covalente (par exemple, la biotine dans des enzymes telles que la pyruvate carboxylase ). [56]
Un exemple d’enzyme qui contient un cofacteur est l’anhydrase carbonique , qui utilise un cofacteur de zinc lié dans le cadre de son site actif. [57] Ces ions ou molécules étroitement liés se trouvent généralement dans le site actif et sont impliqués dans la catalyse. [1] : 8.1.1 Par exemple, les cofacteurs de la flavine et de l’hème sont souvent impliqués dans les réactions redox . [1] : 17
Les enzymes qui nécessitent un cofacteur mais qui n’ont pas de liaison sont appelées apoenzymes ou apoprotéines . Une enzyme avec le ou les cofacteurs nécessaires à l’activité est appelée holoenzyme (ou haloenzyme). Le terme holoenzyme peut également être appliqué aux enzymes qui contiennent plusieurs sous-unités protéiques, telles que les ADN polymérases ; ici l’holoenzyme est le complexe complet contenant toutes les sous-unités nécessaires à l’activité. [1] : 8.1.1
Coenzymes
Les coenzymes sont de petites molécules organiques qui peuvent être liées de manière lâche ou étroite à une enzyme. Les coenzymes transportent des groupes chimiques d’une enzyme à une autre. [58] Les exemples incluent le NADH , le NADPH et l’ adénosine triphosphate (ATP). Certaines coenzymes, telles que la flavine mononucléotide (FMN), la flavine adénine dinucléotide (FAD), le pyrophosphate de thiamine (TPP) et le tétrahydrofolate (THF), sont dérivées de vitamines . Ces coenzymes ne peuvent pas être synthétisées par le corps de novo et des composés étroitement apparentés (vitamines) doivent être acquis à partir de l’alimentation. Les groupes chimiques transportés comprennent :
- l’ ion hydrure (H − ), porté par NAD ou NADP +
- le groupement phosphate, porté par l’ adénosine triphosphate
- le groupement acétyle, porté par la coenzyme A
- des groupements formyle, méthényle ou méthyle, portés par l’acide folique et
- le groupement méthyle, porté par la S-adénosylméthionine [58]
Étant donné que les coenzymes sont chimiquement modifiées à la suite de l’action enzymatique, il est utile de considérer les coenzymes comme une classe spéciale de substrats, ou seconds substrats, qui sont communs à de nombreuses enzymes différentes. Par exemple, environ 1000 enzymes sont connues pour utiliser la coenzyme NADH. [59]
Les coenzymes sont généralement régénérées en continu et leurs concentrations maintenues à un niveau constant à l’intérieur de la cellule. Par exemple, le NADPH est régénéré par la voie du pentose phosphate et la S -adénosylméthionine par la méthionine adénosyltransférase . Cette régénération continue signifie que de petites quantités de coenzymes peuvent être utilisées de manière très intensive. Par exemple, le corps humain transforme chaque jour son propre poids en ATP. [60]
Thermodynamique
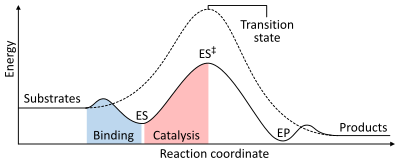
 Les énergies des étapes d’une réaction chimique . Non catalysés (ligne pointillée), les substrats ont besoin de beaucoup d’ énergie d’activation pour atteindre un état de transition , qui se désintègre ensuite en produits à faible énergie. Lorsqu’elle est catalysée par l’enzyme (ligne pleine), l’enzyme lie les substrats (ES), puis stabilise l’état de transition (ES ‡ ) pour réduire l’énergie d’activation nécessaire pour produire des produits (EP) qui sont finalement libérés.
Les énergies des étapes d’une réaction chimique . Non catalysés (ligne pointillée), les substrats ont besoin de beaucoup d’ énergie d’activation pour atteindre un état de transition , qui se désintègre ensuite en produits à faible énergie. Lorsqu’elle est catalysée par l’enzyme (ligne pleine), l’enzyme lie les substrats (ES), puis stabilise l’état de transition (ES ‡ ) pour réduire l’énergie d’activation nécessaire pour produire des produits (EP) qui sont finalement libérés.
Comme avec tous les catalyseurs, les enzymes ne modifient pas la position de l’équilibre chimique de la réaction. En présence d’une enzyme, la réaction se déroule dans le même sens que sans l’enzyme, juste plus rapidement. [1] : 8.2.3 Par exemple, l’anhydrase carbonique catalyse sa réaction dans les deux sens en fonction de la concentration de ses réactifs : [61]
CO 2 + H 2 O → Carbonic anhydrase H 2 CO 3 {displaystyle {ce {CO2{}+H2O->[{text{Anhydrase carbonique}}]H2CO3}}} ![{displaystyle {ce {CO2{}+H2O->[{text{Carbonic anhydrase}}]H2CO3}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/cb4c8837b26e96fe552c17d863f93e0618cd998b) |
( 1 ) |
CO 2 + H 2 O ← Carbonic anhydrase H 2 CO 3 {displaystyle {ce {CO2{}+H2O<-[{text{Anhydrase carbonique}}]H2CO3}}} ![{displaystyle {ce {CO2{}+H2O<-[{text{Carbonic anhydrase}}]H2CO3}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/618e95485aa1c3c44a29c557ac448ae5b544ff07) |
( 2 ) |
La vitesse d’une réaction dépend de l’ énergie d’activation nécessaire pour former l’ état de transition qui se désintègre ensuite en produits. Les enzymes augmentent les taux de réaction en abaissant l’énergie de l’état de transition. Premièrement, la liaison forme un complexe enzyme-substrat (ES) à faible énergie. Deuxièmement, l’enzyme stabilise l’état de transition de sorte qu’il nécessite moins d’énergie pour être atteint par rapport à la réaction non catalysée (ES ‡ ). Enfin, le complexe enzyme-produit (EP) se dissocie pour libérer les produits. [1] : 8,3
Les enzymes peuvent coupler deux réactions ou plus, de sorte qu’une réaction thermodynamiquement favorable peut être utilisée pour “conduire” une réaction thermodynamiquement défavorable de sorte que l’énergie combinée des produits soit inférieure à celle des substrats. Par exemple, l’hydrolyse de l’ ATP est souvent utilisée pour déclencher d’autres Réactions chimiques. [62]
Cinétique

 Un mécanisme de réaction chimique avec ou sans catalyse enzymatique . L’enzyme (E) se lie au substrat (S) pour produire le produit (P).
Un mécanisme de réaction chimique avec ou sans catalyse enzymatique . L’enzyme (E) se lie au substrat (S) pour produire le produit (P). 
 Courbe de saturation pour une réaction enzymatique montrant la relation entre la concentration du substrat et la Vitesse de réaction.
Courbe de saturation pour une réaction enzymatique montrant la relation entre la concentration du substrat et la Vitesse de réaction.
La cinétique enzymatique est l’étude de la manière dont les enzymes se lient aux substrats et les transforment en produits. [63] Les données de taux utilisées dans les analyses cinétiques sont généralement obtenues à partir d’ essais enzymatiques . En 1913 , Leonor Michaelis et Maud Leonora Menten ont proposé une théorie quantitative de la cinétique enzymatique, appelée cinétique de Michaelis-Menten . [64]La contribution majeure de Michaelis et Menten a été de penser aux réactions enzymatiques en deux étapes. Dans le premier, le substrat se lie de manière réversible à l’enzyme, formant le complexe enzyme-substrat. Ceci est parfois appelé le complexe Michaelis-Menten en leur honneur. L’enzyme catalyse ensuite l’étape chimique de la réaction et libère le produit. Ce travail a été développé par GE Briggs et JBS Haldane , qui ont dérivé des équations cinétiques qui sont encore largement utilisées aujourd’hui. [65]
Les taux d’enzymes dépendent des conditions de la solution et de la concentration du substrat . Pour trouver la vitesse maximale d’une réaction enzymatique, la concentration du substrat est augmentée jusqu’à ce qu’une vitesse constante de formation de produit soit observée. Ceci est indiqué dans la courbe de saturation à droite. La saturation se produit parce que, à mesure que la concentration du substrat augmente, de plus en plus d’enzyme libre est convertie en complexe ES lié au substrat. À la Vitesse de réaction maximale ( Vmax ) de l’enzyme, tous les sites actifs de l’enzyme sont liés au substrat et la quantité de complexe ES est la même que la quantité totale d’enzyme. [1] : 8,4
V max n’est qu’un des nombreux paramètres cinétiques importants. La quantité de substrat nécessaire pour obtenir une Vitesse de réaction donnée est également importante. Ceci est donné par la constante de Michaelis-Menten ( K m ), qui est la concentration de substrat requise pour qu’une enzyme atteigne la moitié de sa Vitesse de réaction maximale ; généralement, chaque enzyme a un KM caractéristique pour un substrat donné. Une autre constante utile est k cat , également appelée nombre de rotation , qui est le nombre de molécules de substrat manipulées par un site actif par seconde. [1] : 8,4
L’efficacité d’une enzyme peut être exprimée en termes de k cat / K m . Ceci est également appelé la constante de spécificité et intègre les constantes de vitesse pour toutes les étapes de la réaction jusqu’à et y compris la première étape irréversible. Étant donné que la constante de spécificité reflète à la fois l’affinité et la capacité catalytique, elle est utile pour comparer différentes enzymes les unes par rapport aux autres, ou la même enzyme avec différents substrats. Le maximum théorique de la constante de spécificité est appelé limite de diffusion et est d’environ 10 8 à 10 9 (M −1 s −1). À ce stade, chaque collision de l’enzyme avec son substrat entraînera une catalyse, et la vitesse de formation du produit n’est pas limitée par la Vitesse de réaction mais par la vitesse de diffusion. Les enzymes possédant cette propriété sont appelées catalytiquement parfaites ou cinétiquement parfaites . Des exemples de telles enzymes sont la triose-phosphate isomérase , l’anhydrase carbonique , l’ acétylcholinestérase , la catalase , la fumarase , la β-lactamase et la superoxyde dismutase . [1] : 8.4.2 Le renouvellement de telles enzymes peut atteindre plusieurs millions de réactions par seconde. [1] : 9.2 Mais la plupart des enzymes sont loin d’être parfaites : les valeurs moyennes de k c a t / K m {displaystyle k_{rm {cat}}/K_{rm {m}}} 
et k c a t {displaystyle k_{rm {chat}}}

sont à propos de 10 5 s − 1 M − 1 {displaystyle 10^{5}{rm {s}}^{-1}{rm {M}}^{-1}}

et 10 s − 1 {displaystyle 10{rm {s}}^{-1}}

, respectivement. [66]
La cinétique de Michaelis-Menten repose sur la loi d’action de masse , qui est dérivée des hypothèses de diffusion libre et de collision aléatoire à commande thermodynamique. De nombreux processus biochimiques ou cellulaires s’écartent considérablement de ces conditions, en raison de l’ encombrement macromoléculaire et du mouvement moléculaire contraint. [67] Des extensions plus récentes et complexes du modèle tentent de corriger ces effets. [68]
Inhibition
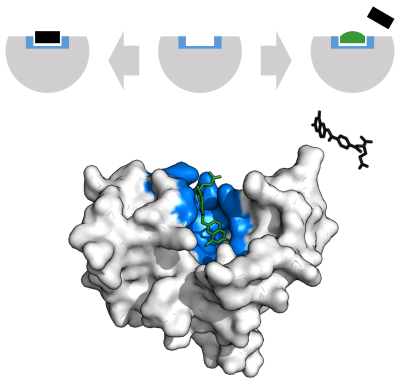


 Le coenzyme acide folique (à gauche) et le médicament anticancéreux méthotrexate (à droite) ont une structure très similaire (les différences apparaissent en vert). En conséquence, le méthotrexate est un inhibiteur compétitif de nombreuses enzymes qui utilisent les folates.
Le coenzyme acide folique (à gauche) et le médicament anticancéreux méthotrexate (à droite) ont une structure très similaire (les différences apparaissent en vert). En conséquence, le méthotrexate est un inhibiteur compétitif de nombreuses enzymes qui utilisent les folates.
Les taux de réaction enzymatique peuvent être diminués par divers types d’ inhibiteurs enzymatiques . [69] : 73–74
Types d’inhibition
Compétitif
Un inhibiteur compétitif et un substrat ne peuvent pas se lier à l’enzyme en même temps. [70] Souvent, les inhibiteurs compétitifs ressemblent fortement au substrat réel de l’enzyme. Par exemple, le médicament méthotrexate est un inhibiteur compétitif de l’enzyme dihydrofolate réductase , qui catalyse la réduction du dihydrofolate en tétrahydrofolate. [71] La similitude entre les structures du dihydrofolate et de ce médicament est illustrée dans la figure ci-jointe. Ce type d’inhibition peut être surmonté avec une forte concentration de substrat. Dans certains cas, l’inhibiteur peut se lier à un site autre que le site de liaison du substrat habituel et exercer un effet allostériquemodifier la forme du site de liaison habituel. [72]
Non compétitif
Un inhibiteur non compétitif se lie à un site autre que celui où le substrat se lie. Le substrat se lie toujours avec son affinité habituelle et donc Km reste le même. Cependant, l’inhibiteur réduit l’efficacité catalytique de l’enzyme de sorte que la Vmax est réduite. Contrairement à l’inhibition compétitive, l’inhibition non compétitive ne peut pas être surmontée avec une concentration élevée de substrat. [69] : 76–78
Non compétitif
Un inhibiteur non compétitif ne peut pas se lier à l’enzyme libre, uniquement au complexe enzyme-substrat ; par conséquent, ces types d’inhibiteurs sont plus efficaces à une concentration de substrat élevée. En présence de l’inhibiteur, le complexe enzyme-substrat est inactif. [69] : 78 Ce type d’inhibition est rare. [73]
Mixte
Un inhibiteur mixte se lie à un Site allostérique et la liaison du substrat et de l’inhibiteur s’influence mutuellement. La fonction de l’enzyme est réduite mais pas éliminée lorsqu’elle est liée à l’inhibiteur. Ce type d’inhibiteur ne suit pas l’équation de Michaelis-Menten. [69] : 76–78
Irréversible
Un inhibiteur irréversible inactive définitivement l’enzyme, généralement en formant une liaison covalente avec la protéine. [74] La pénicilline [75] et l’aspirine [76] sont des médicaments courants qui agissent de cette manière.
Fonctions des inhibiteurs
Dans de nombreux organismes, les inhibiteurs peuvent agir dans le cadre d’un mécanisme de rétroaction . Si une enzyme produit trop d’une substance dans l’organisme, cette substance peut agir comme un inhibiteur de l’enzyme au début de la voie qui la produit, provoquant un ralentissement ou un arrêt de la production de la substance lorsqu’il y en a une quantité suffisante. Il s’agit d’une forme de rétroaction négative . Des voies métaboliques majeures telles que le cycle de l’acide citrique utilisent ce mécanisme. [1] : 17.2.2
Étant donné que les inhibiteurs modulent la fonction des enzymes, ils sont souvent utilisés comme médicaments. Beaucoup de ces médicaments sont des inhibiteurs compétitifs réversibles qui ressemblent au substrat natif de l’enzyme, similaire au méthotrexate ci-dessus; d’autres exemples bien connus incluent les statines utilisées pour traiter l’ hypercholestérolémie [ 77] et les inhibiteurs de protéase utilisés pour traiter les infections rétrovirales telles que le VIH . [78] Un exemple courant d’inhibiteur irréversible utilisé comme médicament est l’aspirine , qui inhibe les enzymes COX-1 et COX-2 qui produisent le messager de l’ inflammation .prostaglandine . [76] D’autres inhibiteurs d’enzymes sont des poisons. Par exemple, le poison cyanure est un inhibiteur enzymatique irréversible qui se combine avec le cuivre et le fer dans le site actif de l’enzyme cytochrome c oxydase et bloque la respiration cellulaire . [79]
Facteurs affectant l’activité enzymatique
Comme les enzymes sont constituées de protéines, leurs actions sont sensibles aux changements de nombreux facteurs physiochimiques tels que le pH, la température, la concentration du substrat, etc.
Le tableau suivant montre les optima de pH pour diverses enzymes. [80]
| Enzyme | pH optimal | Descriptif pH |
|---|---|---|
| Pepsine | 1,5–1,6 | Très acide |
| Invertase | 4.5 | Acide |
| Lipase (estomac) | 4.0–5.0 | Acide |
| Lipase (huile de ricin) | 4.7 | Acide |
| Lipase (pancréas) | 8.0 | Alcalin |
| Amylase (malt) | 4.6–5.2 | Acide |
| Amylase (pancréas) | 6,7–7,0 | Acide-neutre |
| Cellobiase | 5.0 | Acide |
| maltais | 6.1–6.8 | Acide |
| Sucrase | 6.2 | Acide |
| Catalase | 7.0 | Neutre |
| Uréase | 7.0 | Neutre |
| Cholinestérase | 7.0 | Neutre |
| Ribonucléase | 7,0–7,5 | Neutre |
| Fumarase | 7.8 | Alcalin |
| Trypsine | 7.8–8.7 | Alcalin |
| L’adénosine triphosphate | 9.0 | Alcalin |
| Arginase | 10.0 | Très alcalin |
Fonction Biologique
Les enzymes remplissent une grande variété de fonctions à l’ intérieur des organismes vivants. Ils sont indispensables à la transduction du signal et à la régulation cellulaire, souvent via les kinases et les phosphatases . [81] Ils génèrent également du mouvement, la myosine hydrolysant l’ATP pour générer une contraction musculaire , et transportent également la cargaison autour de la cellule dans le cadre du cytosquelette . [82] D’autres ATPases dans la membrane cellulaire sont des pompes ioniques impliquées dans le transport actif . Les enzymes sont également impliquées dans des fonctions plus exotiques, telles que la luciférase générant de la lumière chez les lucioles. [83] Les virus peuvent également contenir des enzymes pour infecter les cellules, telles que l’ intégrase du VIH et la transcriptase inverse , ou pour la libération virale des cellules, comme la neuraminidase du virus de la grippe . [84]
Une fonction importante des enzymes est dans le système digestif des animaux. Les enzymes telles que les amylases et les protéases décomposent les grosses molécules ( amidon ou protéines , respectivement) en plus petites, afin qu’elles puissent être absorbées par les intestins. Les molécules d’amidon, par exemple, sont trop grosses pour être absorbées par l’intestin, mais les enzymes hydrolysent les chaînes d’amidon en molécules plus petites telles que le maltose et éventuellement le glucose , qui peuvent ensuite être absorbées. Différentes enzymes digèrent différentes substances alimentaires. Chez les ruminants , qui ont un régime herbivore , les micro-organismes de l’intestin produisent une autre enzyme,cellulase , pour décomposer les parois cellulaires cellulosiques des fibres végétales. [85]
Métabolisme

 La voie métabolique de la glycolyse libère de l’énergie en convertissant le glucose en pyruvate via une série de métabolites intermédiaires. Chaque modification chimique (encadré rouge) est effectuée par une enzyme différente.
La voie métabolique de la glycolyse libère de l’énergie en convertissant le glucose en pyruvate via une série de métabolites intermédiaires. Chaque modification chimique (encadré rouge) est effectuée par une enzyme différente.
Plusieurs enzymes peuvent travailler ensemble dans un ordre spécifique, créant des voies métaboliques . [1] : 30.1 Dans une voie métabolique, une enzyme prend le produit d’une autre enzyme comme substrat. Après la réaction catalytique, le produit est ensuite transmis à une autre enzyme. Parfois, plus d’une enzyme peut catalyser la même réaction en parallèle ; cela peut permettre une régulation plus complexe : avec, par exemple, une faible activité constante apportée par une enzyme mais une forte activité inductible par une seconde enzyme. [86]
Les enzymes déterminent les étapes qui se produisent dans ces voies. Sans enzymes, le métabolisme ne progresserait pas par les mêmes étapes et ne pourrait pas être régulé pour répondre aux besoins de la cellule. La plupart des voies métaboliques centrales sont régulées à quelques étapes clés, généralement par des enzymes dont l’activité implique l’hydrolyse de l’ ATP . Parce que cette réaction libère tellement d’énergie, d’autres réactions thermodynamiquement défavorables peuvent être couplées à l’hydrolyse de l’ATP, entraînant la série globale de réactions métaboliques liées. [1] : 30,1
Contrôle de l’activité
Il existe cinq façons principales de contrôler l’activité enzymatique dans la cellule. [1] : 30.1.1
Régulation
Les enzymes peuvent être activées ou inhibées par d’autres molécules. Par exemple, le ou les produits finaux d’une voie métabolique sont souvent des inhibiteurs de l’une des premières enzymes de la voie (généralement la première étape irréversible, appelée étape engagée), régulant ainsi la quantité de produit final fabriquée par les voies. Un tel mécanisme de régulation est appelé mécanisme de rétroaction négative , car la quantité de produit final produit est régulée par sa propre concentration. [87] : 141–48 Le mécanisme de rétroaction négative peut ajuster efficacement le taux de synthèse des métabolites intermédiaires en fonction des demandes des cellules. Cela contribue à une allocation efficace des matériaux et à l’économie d’énergie, et empêche la fabrication excessive de produits finis. Comme d’autres dispositifs homéostatiques , le contrôle de l’action enzymatique permet de maintenir un environnement interne stable dans les organismes vivants. [87] : 141
Modification post-traductionnelle
Des exemples de modification post-traductionnelle comprennent la phosphorylation , la myristoylation et la glycosylation . [87] : 149–69 Par exemple, dans la réponse à l’insuline , la phosphorylation de plusieurs enzymes, dont la glycogène synthase , aide à contrôler la synthèse ou la dégradation du glycogène et permet à la cellule de répondre aux variations de la glycémie . [88] Un autre exemple de modification post-traductionnelle est le clivage de la chaîne polypeptidique. La chymotrypsine , une protéase digestive, est produit sous forme inactive sous forme de chymotrypsinogène dans le pancréas et transporté sous cette forme jusqu’à l’ estomac où il est activé. Cela empêche l’enzyme de digérer le pancréas ou d’autres tissus avant qu’il ne pénètre dans l’intestin. Ce type de précurseur inactif d’une enzyme est connu sous le nom de zymogène [87] : 149–53 ou proenzyme.
Quantité
La production d’enzymes ( transcription et traduction de gènes d’enzymes) peut être augmentée ou diminuée par une cellule en réponse à des changements dans l’environnement de la cellule. Cette forme de régulation génique est appelée induction enzymatique . Par exemple, les bactéries peuvent devenir résistantes aux Antibiotiques tels que la pénicilline parce que des enzymes appelées bêta-lactamases sont induites qui hydrolysent l’ anneau bêta-lactame crucial dans la molécule de pénicilline. [89] Un autre exemple provient des enzymes du foie appelées cytochrome P450 oxydases , qui jouent un rôle important dans le métabolisme des médicaments .. L’induction ou l’inhibition de ces enzymes peut provoquer des interactions médicamenteuses . [90] Les niveaux d’enzymes peuvent également être régulés en modifiant le taux de dégradation enzymatique . [1] : 30.1.1 Le contraire de l’induction enzymatique est la répression enzymatique .
Distribution subcellulaire
Les enzymes peuvent être compartimentées, avec différentes voies métaboliques se produisant dans différents compartiments cellulaires . Par exemple, les acides gras sont synthétisés par un ensemble d’enzymes dans le cytosol , le réticulum endoplasmique et l’appareil de Golgi et utilisés par un ensemble différent d’enzymes comme source d’énergie dans la mitochondrie , par β-Oxydation . [91] De plus, le trafic de l’enzyme vers différents compartiments peut modifier le degré de protonation (par exemple, le cytoplasme neutre et le lysosome acide ) ou l’état oxydatif (par exemple, l’oxydationpériplasme ou cytoplasme réducteur ) qui à son tour affecte l’activité enzymatique. [92] Contrairement à la partition en organelles liées à la membrane, la localisation subcellulaire de l’enzyme peut également être modifiée par la polymérisation d’enzymes dans des filaments cytoplasmiques macromoléculaires. [93] [94]
Spécialisation d’orgue
Chez les eucaryotes multicellulaires , les cellules de différents organes et tissus ont différents modèles d’ expression génique et ont donc différents ensembles d’enzymes (appelés isozymes ) disponibles pour les réactions métaboliques. Cela fournit un mécanisme de régulation du métabolisme global de l’organisme. Par exemple, l’ hexokinase , la première enzyme de la voie de la glycolyse , a une forme spécialisée appelée glucokinase exprimée dans le foie et le pancréas qui a une affinité plus faible pour le glucose mais est plus sensible à la concentration de glucose. [95]Cette enzyme est impliquée dans la détection de la glycémie et la régulation de la production d’ insuline . [96]
Implication dans la maladie

 Dans la phénylalanine hydroxylase , plus de 300 mutations différentes dans toute la structure provoquent la phénylcétonurie . Substrat phénylalanine et coenzyme tétrahydrobioptérine en noir et cofacteur Fe 2+ en jaune. ( APB : 1KW0 )
Dans la phénylalanine hydroxylase , plus de 300 mutations différentes dans toute la structure provoquent la phénylcétonurie . Substrat phénylalanine et coenzyme tétrahydrobioptérine en noir et cofacteur Fe 2+ en jaune. ( APB : 1KW0 ) 
 Les défauts héréditaires des enzymes sont généralement hérités de manière autosomique car il y a plus de chromosomes non X que de chromosomes X, et de manière récessive car les enzymes des gènes non affectés sont généralement suffisantes pour prévenir les symptômes chez les porteurs.
Les défauts héréditaires des enzymes sont généralement hérités de manière autosomique car il y a plus de chromosomes non X que de chromosomes X, et de manière récessive car les enzymes des gènes non affectés sont généralement suffisantes pour prévenir les symptômes chez les porteurs.
Le contrôle strict de l’activité enzymatique étant essentiel à l’ homéostasie , tout dysfonctionnement (mutation, surproduction, sous-production ou délétion) d’une seule enzyme critique peut entraîner une maladie génétique . Le dysfonctionnement d’un seul type d’enzyme parmi les milliers de types présents dans le corps humain peut être fatal. Un exemple de maladie génétique mortelle due à une insuffisance enzymatique est la maladie de Tay-Sachs , dans laquelle les patients manquent de l’enzyme hexosaminidase . [97] [98]
Un exemple de déficit enzymatique est le type le plus courant de phénylcétonurie . De nombreuses mutations différentes d’un seul acide aminé dans l’enzyme phénylalanine hydroxylase , qui catalyse la première étape de la dégradation de la phénylalanine , entraînent une accumulation de phénylalanine et de produits apparentés. Certaines mutations se trouvent dans le site actif, perturbant directement la liaison et la catalyse, mais beaucoup sont loin du site actif et réduisent l’activité en déstabilisant la structure de la protéine ou en affectant une oligomérisation correcte. [99] [100] Cela peut conduire à une déficience intellectuelle si la maladie n’est pas traitée. [101] Un autre exemple est le déficit en pseudocholinestérase, dans lequel la capacité du corps à décomposer les médicaments à base d’ester de choline est altérée. [102] L’administration orale d’enzymes peut être utilisée pour traiter certaines déficiences enzymatiques fonctionnelles, telles que l’insuffisance pancréatique [103] et l’intolérance au lactose . [104]
Une autre façon dont les dysfonctionnements enzymatiques peuvent provoquer des maladies provient de mutations germinales dans les gènes codant pour les enzymes de réparation de l’ADN . Les défauts de ces enzymes causent le cancer parce que les cellules sont moins capables de réparer les mutations dans leurs génomes . Cela provoque une lente accumulation de mutations et entraîne le développement de cancers . Un exemple d’un tel syndrome de cancer héréditaire est le xeroderma pigmentosum , qui provoque le développement de cancers de la peau en réponse à une exposition même minime à la lumière ultraviolette . [105] [106]
Évolution
Semblables à toute autre protéine, les enzymes changent avec le temps par le biais de mutations et de divergences de séquence. Compte tenu de leur rôle central dans le métabolisme , l’évolution des enzymes joue un rôle critique dans l’ adaptation . Une question clé est donc de savoir si et comment les enzymes peuvent modifier leurs activités enzymatiques parallèlement. Il est généralement admis que de nombreuses nouvelles activités enzymatiques ont évolué par duplication de gènes et mutation des copies en double, bien que l’évolution puisse également se produire sans duplication. Un exemple d’enzyme qui a changé son activité est l’ancêtre de la méthionyl amino peptidase (MAP) et de la créatine amidinohydrolase ( créatinase) qui sont clairement homologues mais catalysent des réactions très différentes (MAP élimine la méthionine amino-terminale dans les nouvelles protéines tandis que la créatinase hydrolyse la créatine en sarcosine et urée ). De plus, la MAP dépend des ions métalliques alors que la créatinase ne l’est pas, par conséquent cette propriété a également été perdue avec le temps. [107] De petits changements d’activité enzymatique sont extrêmement courants parmi les enzymes. En particulier, la spécificité de liaison au substrat (voir ci-dessus) peut facilement et rapidement changer avec des changements d’acides aminés simples dans leurs poches de liaison au substrat. Ceci est fréquemment observé dans les principales classes d’enzymes telles que les kinases . [108]
L’évolution artificielle (in vitro) est maintenant couramment utilisée pour modifier l’activité ou la spécificité enzymatique pour des applications industrielles (voir ci-dessous).
Applications industrielles
Les enzymes sont utilisées dans l’ industrie chimique et d’autres applications industrielles lorsque des catalyseurs extrêmement spécifiques sont nécessaires. Les enzymes en général sont limitées dans le nombre de réactions qu’elles ont évolué pour catalyser et aussi par leur manque de stabilité dans les solvants organiques et à des températures élevées. Par conséquent, l’ingénierie des protéines est un domaine de recherche actif et implique des tentatives de création de nouvelles enzymes dotées de nouvelles propriétés, soit par une conception rationnelle, soit par une évolution in vitro . [109] [110] Ces efforts ont commencé à être couronnés de succès et quelques enzymes ont maintenant été conçues “à partir de zéro” pour catalyser des réactions qui ne se produisent pas dans la nature. [111]
| Application | Enzymes utilisées | Les usages |
|---|---|---|
| Industrie des biocarburants | Cellulases | Décomposer la cellulose en sucres qui peuvent être fermentés pour produire de l’ éthanol cellulosique . [112] |
| Ligninases | Prétraitement de la biomasse pour la production de biocarburant. [112] | |
| Détergent Biologique | Protéases , amylases , lipases | Enlevez les taches de protéines, d’amidon et de graisse ou d’huile du linge et de la vaisselle. [113] |
| Mannanases | Enlevez les taches de nourriture de l’additif alimentaire commun gomme de guar . [113] | |
| Industrie brassicole | Amylase , glucanases , protéases | Séparation des polysaccharides et des protéines dans le malt . [114] : 150–9 |
| Bêtaglucanases | Améliorer les caractéristiques de filtration du moût et de la bière. [114] : 545 | |
| Amyloglucosidase et pullulanases | Faites de la bière à faible teneur en calories et ajustez la fermentescibilité. [114] : 575 | |
| Acétolactate décarboxylase (ALDC) | Augmenter l’efficacité de la fermentation en réduisant la formation de diacétyle . [115] | |
| Utilisations culinaires | Papaïne | Attendrir la viande pour la cuisson. [116] |
| Industrie laitière | Rénine | Hydrolyser les protéines dans la fabrication du fromage . [117] |
| Lipases | Produire du camembert et des fromages persillés comme le roquefort . [118] | |
| Préparation des aliments | Amylases | Produire des sucres à partir d’ amidon , comme dans la fabrication de sirop de maïs à haute teneur en fructose . [119] |
| Protéases | Baisser le taux de protéines de la farine , comme en biscuiterie . [120] | |
| Trypsine | Fabriquer des aliments hypoallergéniques pour bébés. [120] | |
| Cellulases , pectinases | Clarifier les jus de fruits . [121] | |
| Biologie moléculaire | Nucléases , ADN ligase et polymérases | Utilisez la digestion de restriction et la réaction en chaîne par polymérase pour créer de l’ADN recombinant . [1] : 6,2 |
| Industrie du papier | Xylanases , hémicellulases et lignine peroxydases | Retirer la lignine de la pâte kraft . [122] |
| Soins personnels | Protéases | Enlevez les protéines sur les lentilles de contact pour prévenir les infections. [123] |
| Industrie de l’amidon | Amylases | Convertit l’amidon en glucose et divers sirops . [124] |
Voir également
-
![icon]()
![icon]() Portail de biologie
Portail de biologie -
![icon]()
![icon]() Portail alimentaire
Portail alimentaire
- Enzymes industrielles
- Liste des enzymes
- Machine moléculaire
Bases de données enzymatiques
- BRENDA
- ExPASy
- IntEnz
- KEGG
- MetaCyc
Références
- ^ un bcd e f g h i j k l m n o p q r s t u Stryer L , Berg JM, Tymoczko JL (2002). Biochimie (5e éd.). San Francisco : WH Freeman. ISBN 0-7167-4955-6.
![open access]()
- ^ Murphy JM, Farhan H, Eyers PA (2017). “Bio-Zombie: la montée des pseudoenzymes en biologie”. Biochem Soc Trans . 45 (2): 537–544. doi : 10.1042/bst20160400 . PMID 28408493 .
- ^ Murphy JM, et al. (2014). “Une méthodologie robuste pour sous-classifier les pseudokinases en fonction de leurs propriétés de liaison aux nucléotides” . Journal biochimique . 457 (2): 323–334. doi : 10.1042/BJ20131174 . PMC 5679212 . PMID 24107129 .
- ^ Schomburg I, Chang A, Placzek S, Söhngen C, Rother M, Lang M, Munaretto C, Ulas S, Stelzer M, Grote A, Scheer M, Schomburg D (janvier 2013). “BRENDA en 2013 : réactions intégrées, données cinétiques, données sur la fonction enzymatique, classification améliorée des maladies : nouvelles options et contenus dans BRENDA” . Recherche sur les acides nucléiques . 41 (numéro de base de données) : D764–72. doi : 10.1093/nar/gks1049 . PMC 3531171 . PMID 23203881 .
- ^ Radzicka A, Wolfenden R (janvier 1995). “Une enzyme compétente”. Sciences . 267 (5194): 90–931. Bibcode : 1995Sci…267…90R . doi : 10.1126/science.7809611 . PMID 7809611 . S2CID 8145198 .
- ^ Callahan BP, Miller BG (décembre 2007). “OMP décarboxylase – Une énigme persiste”. Chimie bioorganique . 35 (6) : 465–9. doi : 10.1016/j.bioorg.2007.07.004 . PMID 17889251 .
- ^ de Réaumur RA (1752). “Observations sur la digestion des oiseaux”. Histoire de l’Académie Royale des Sciences . 1752 : 266, 461.
- ^ Williams HS (1904). Une histoire des sciences : en cinq tomes . Volume IV : Développement moderne des sciences chimiques et biologiques . Harper et frères.
- ^ Payen A, Persoz JF (1833). “Mémoire sur la diastase, les principaux produits de ses réactions et leurs applications aux arts industriels” [Mémoire sur la diastase, les principaux produits de ses réactions et leurs applications aux arts industriels]. Annales de chimie et de physique . 2e (en français). 53 : 73–92.
- ^ Manchester KL (décembre 1995). “Louis Pasteur (1822–1895) – le hasard et l’esprit préparé”. Tendances en biotechnologie . 13 (12): 511–5. doi : 10.1016/S0167-7799(00)89014-9 . PMID 8595136 .
- ↑ Kühne a inventé le mot « enzyme » dans : Kühne W (1877). “Über das Verhalten verschiedener organisirter und sog. ungeformter Fermente” [Sur le comportement de divers ferments organisés et dits informes]. Verhandlungen des Naturhistorisch-medicinischen Vereins zu Heidelberg . nouvelle série (en allemand). 1 (3): 190–193.Passage pertinent de la page 190 : « Um Missverständnissen vorzubeugen und lästige Umschreibungen zu vermeiden schlägt Vortragender vor, die ungeformten oder nicht organisirten Fermente, deren Wirkung ohne Anwesenheit von Organismen und ausserhalb derselben erfolgen kann, als Enzyme zu bezeichnen. (Traduction : Afin de dissiper les malentendus et d’éviter les périphrases encombrantes, [l’auteur, professeur d’université] propose de désigner comme “enzymes” les ferments non formés ou non organisés, dont l’action peut se produire sans la présence d’organismes et en dehors de ceux-ci.)
- ^ Holmes FL (2003). “Enzymes” . Dans Heilbron JL (éd.). Le compagnon d’Oxford à l’histoire de la science moderne . Oxford : presse universitaire d’Oxford. p. 270. ISBN 9780199743766.
- ^ “Eduard Büchner” . Biographie du lauréat du prix Nobel . Nobelprize.org . Récupéré le 23 février 2015 .
- ^ “Eduard Buchner – Conférence Nobel: Fermentation sans cellule” . Nobelprize.org . 1907 . Récupéré le 23 février 2015 .
- ^ La dénomination des enzymes en ajoutant le suffixe “-ase” au substrat sur lequel l’enzyme agit, a été attribuée au scientifique français Émile Duclaux (1840-1904), qui avait l’intention d’honorer les découvreurs de la diastase – la première enzyme à être isolé – en introduisant cette pratique dans son livre Duclaux E (1899). Traité de microbiologie : Diastases , toxines et venins . Paris, France : Masson et Cie.Voir le chapitre 1, en particulier la page 9.
- ^ Willstatter R (1927). “Conférence Faraday. Problèmes et méthodes de recherche sur les enzymes”. Journal of the Chemical Society (reprise) : 1359–1381. doi : 10.1039/JR9270001359 . cité dans Blow D (avril 2000). « Alors, comprenons-nous comment fonctionnent les enzymes ? » . Structure . 8 (4) : R77–R81. doi : 10.1016/S0969-2126(00)00125-8 . PMID 10801479 .
- ^ “Prix Nobel et Lauréats : Le Prix Nobel de Chimie 1946” . Nobelprize.org . Récupéré le 23 février 2015 .
- ^ Blake CC, Koenig DF, Mair GA, North AC, Phillips DC, Sarma VR (mai 1965). “Structure du lysozyme de blanc d’oeuf de poule. Une synthèse de Fourier tridimensionnelle à une résolution de 2 Ångström”. Nature . 206 (4986): 757–61. Bibcode : 1965Natur.206..757B . doi : 10.1038/206757a0 . PMID 5891407 . S2CID 4161467 .
- ^ Johnson LN, Petsko GA (1999). “David Phillips et l’origine de l’enzymologie structurale”. Tendances Biochem. Sci . 24 (7): 287–9. doi : 10.1016/S0968-0004(99)01423-1 . PMID 10390620 .
- ^ Moss, généraliste. “Recommandations du Comité de Nomenclature de l’Union Internationale de Biochimie et de Biologie Moléculaire sur la Nomenclature et la Classification des Enzymes par les Réactions qu’elles Catalysent” . Union internationale de biochimie et de biologie moléculaire . Récupéré le 28 août 2021 .
- ^ Comité de nomenclature. “CE 2.7.1.1” . Union internationale de biochimie et de biologie moléculaire (NC-IUBMB) . École des sciences biologiques et chimiques, Queen Mary, Université de Londres. Archivé de l’original le 1er décembre 2014 . Récupéré le 6 mars 2015 .
- ^ Mulder NJ (28 septembre 2007). “Bases de données de famille de protéines”. eLS . Chichester, Royaume-Uni : John Wiley & Sons, Ltd. pp. a0003058.pub2. doi : 10.1002/9780470015902.a0003058.pub2 . ISBN 978-0-470-01617-6.
- ^ Anfinsen CB (juillet 1973). “Principes qui régissent le repliement des chaînes protéiques”. Sciences . 181 (4096): 223-230. Bibcode : 1973Sci…181..223A . doi : 10.1126/science.181.4096.223 . PMID 4124164 .
- ^ Dunaway-Mariano D (novembre 2008). “Découverte de la fonction enzymatique” . Structure . 16 (11): 1599–600. doi : 10.1016/j.str.2008.10.001 . PMID 19000810 .
- ^ Petsko GA, Ringe D (2003). “Chapitre 1 : De la séquence à la structure” . Structure et fonction des protéines . Londres : Nouvelle Science. p. 27. ISBN 978-1405119221.
- ^ Chen LH, Kenyon GL, Curtin F, Harayama S, Bembenek ME, Hajipour G, Whitman CP (septembre 1992). “4-Oxalocrotonate tautomérase, une enzyme composée de 62 résidus d’acides aminés par monomère” . Le Journal de chimie Biologique . 267 (25): 17716–21. doi : 10.1016/S0021-9258(19)37101-7 . PMID 1339435 .
- ^ Smith S (décembre 1994). “La synthase d’acide gras animal: un gène, un polypeptide, sept enzymes”. Revue FASEB . 8 (15): 1248–1259. doi : 10.1096/fasebj.8.15.8001737 . PMID 8001737 . S2CID 22853095 .
- ^ “L’Atlas de Site Catalytique” . L’Institut Européen de Bioinformatique . Récupéré le 4 avril 2007 .
- ^ un b Suzuki H (2015). “Chapitre 7 : Structure du site actif”. Comment fonctionnent les enzymes : de la structure à la fonction . Boca Raton, Floride : CRC Press. p. 117–140. ISBN 978-981-4463-92-8.
- ^ Krauss G (2003). “Les réglementations de l’activité enzymatique” . Biochimie de la transduction et de la régulation du signal (3e éd.). Weinheim : Wiley-VCH. p. 89–114. ISBN 9783527605767.
- ^ Jaeger KE, Eggert T (août 2004). “Biocatalyse énantiosélective optimisée par évolution dirigée”. Opinion actuelle en biotechnologie . 15 (4): 305–313. doi : 10.1016/j.copbio.2004.06.007 . PMID 15358000 .
- ^ Shevelev IV, Hübscher U (mai 2002). “Les exonucléases 3′ 5′”. Examens de la nature Biologie cellulaire moléculaire . 3 (5): 364–76. doi : 10.1038/nrm804 . PMID 11988770 . S2CID 31605786 .
- ^ Zenkin N, Yuzenkova Y, Severinov K (juillet 2006). “Relecture transcriptionnelle assistée par transcription”. Sciences . 313 (5786): 518–20. Bibcode : 2006Sci…313..518Z . doi : 10.1126/science.1127422 . PMID 16873663 . S2CID 40772789 .
- ^ Ibba M, Soll D (2000). “Synthèse d’aminoacyl-ARNt”. Revue annuelle de biochimie . 69 : 617–50. doi : 10.1146/annurev.biochem.69.1.617 . PMID 10966471 .
- ^ Rodnina MV, Wintermeyer W (2001). “Fidélité de la sélection des aminoacyl-ARNt sur le ribosome: mécanismes cinétiques et structurels”. Revue annuelle de biochimie . 70 : 415–35. doi : 10.1146/annurev.biochem.70.1.415 . PMID 11395413 .
- ^ Khersonsky O, Tawfik DS (2010). “La promiscuité enzymatique: une perspective mécaniste et évolutive”. Revue annuelle de biochimie . 79 : 471–505. doi : 10.1146/annurev-biochem-030409-143718 . PMID 20235827 .
- ^ O’Brien PJ, Herschlag D (avril 1999). “La promiscuité catalytique et l’évolution de nouvelles activités enzymatiques” . Chimie & Biologie . 6 (4) : R91–R105. doi : 10.1016/S1074-5521(99)80033-7 . PMID 10099128 .
- ^ Fischer E (1894). “Einfluss der Configuration auf die Wirkung der Enzyme” [Influence de la configuration sur l’action des enzymes]. Berichte der Deutschen Chemischen Gesellschaft zu Berlin (en allemand). 27 (3): 2985–93. doi : 10.1002/cber.18940270364 . À partir de la page 2992 : “Um ein Bild zu gebrauchen, will ich sagen, dass Enzym und Glucosid wie Schloss und Schlüssel zu einander passen müssen, um eine chemische Wirkung auf einander ausüben zu können.” (Pour utiliser une image, je dirai qu’une enzyme et un glucoside [c’est-à-dire un dérivé du glucose] doivent s’adapter comme une serrure et une clé, afin de pouvoir exercer un effet chimique l’un sur l’autre.)
- ^ Cooper GM (2000). “Chapitre 2.2 : Le rôle central des enzymes en tant que catalyseurs biologiques” . La cellule: une approche moléculaire (2e éd.). Washington (DC) : ASM Press. ISBN 0-87893-106-6.
- ^ Koshland DE (février 1958). « Application d’une théorie de la spécificité enzymatique à la synthèse des protéines » . Actes de l’Académie nationale des sciences des États-Unis d’Amérique . 44 (2): 98–104. Bibcode : 1958PNAS…44…98K . doi : 10.1073/pnas.44.2.98 . PMC 335371 . PMID 16590179 .
- ^ Vasella A, Davies GJ, Böhm M (octobre 2002). “Mécanismes de la glycosidase”. Opinion actuelle en biologie chimique . 6 (5): 619–629. doi : 10.1016/S1367-5931(02)00380-0 . PMID 12413546 .
- ^ Boyer R (2002). “Chapitre 6: Enzymes I, réactions, cinétique et inhibition”. Concepts en biochimie (2e éd.). New York, Chichester, Weinheim, Brisbane, Singapour, Toronto. : John Wiley & Sons, Inc. pp. 137–8. ISBN 0-470-00379-0. OCLC 51720783 .
- ^ Savir Y, Tlusty T (2007). Scalas E (éd.). « Relecture conformationnelle : l’impact des changements conformationnels sur la spécificité de la reconnaissance moléculaire » . PLOS ONE . 2 (5) : e468. Bibcode : 2007PLoSO…2..468S . doi : 10.1371/journal.pone.0000468 . PMC 1868595 . PMID 17520027 .
- ^ Fersht A (1985). Structure et mécanisme enzymatiques . San Francisco : WH Freeman. p. 50–2. ISBN 978-0-7167-1615-0.
- ^ Warshel A, Sharma PK, Kato M, Xiang Y, Liu H, Olsson MH (août 2006). “Base électrostatique pour la catalyse enzymatique”. Examens chimiques . 106 (8): 3210–35. doi : 10.1021/cr0503106 . PMID 16895325 .
- ^ Cox MM, Nelson DL (2013). “Chapitre 6.2: Comment fonctionnent les enzymes”. Principes de biochimie de Lehninger (6e éd.). New York, NY : WH Freeman. p. 195. ISBN 978-1464109621.
- ^ Benkovic SJ, Hammes-Schiffer S (août 2003). “Une perspective sur la catalyse enzymatique”. Sciences . 301 (5637): 1196–202. Bibcode : 2003Sci…301.1196B . doi : 10.1126/science.1085515 . PMID 12947189 . S2CID 7899320 .
- ^ Jencks WP (1987). Catalyse en Chimie et Enzymologie . Mineola, NY : Douvres. ISBN 978-0-486-65460-7.
- ^ Villa J, Strajbl M, Glennon TM, Sham YY, Chu ZT, Warshel A (octobre 2000). “Quelle est l’importance des contributions entropiques à la catalyse enzymatique ?” . Actes de l’Académie nationale des sciences des États-Unis d’Amérique . 97 (22): 11899–904. Bib code : 2000PNAS …9711899V . doi : 10.1073/pnas.97.22.11899 . PMC 17266 . PMID 11050223 .
- ^ Polgar, L. (7 juillet 2005). “La triade catalytique des sérine peptidases”. Sciences de la vie cellulaire et moléculaire . 62 (19–20): 2161–2172. doi : 10.1007/s00018-005-5160-x . ISSN 1420-682X . PMID 16003488 . S2CID 3343824 .
- ^ Ramanathan A, Savol A, Burger V, Chennubhotla CS, Agarwal PK (2014). « Populations conformationnelles de protéines et sous-états fonctionnellement pertinents ». Acc. Chim. Rés . 47 (1): 149–56. doi : 10.1021/ar400084s . OSTI 1565147 . PMID 23988159 .
- ^ Tsai CJ, Del Sol A, Nussinov R (2009). “Allosterie des protéines, transmission du signal et dynamique: un schéma de classification des mécanismes allostériques” (PDF) . Mol Biosyst . 5 (3): 207-216. doi : 10.1039/b819720b . PMC 2898650 . PMID 19225609 .
- ^ Changeux JP, Edelstein SJ (juin 2005). “Mécanismes allostériques de transduction du signal”. Sciences . 308 (5727) : 1424–8. Bibcode : 2005Sci…308.1424C . doi : 10.1126/science.1108595 . PMID 15933191 . S2CID 10621930 .
- ^ de Bolster MW (1997). “Glossaire des termes utilisés en chimie bioinorganique: cofacteur” . Union internationale de chimie pure et appliquée. Archivé de l’original le 21 janvier 2017 . Récupéré le 30 octobre 2007 .
- ^ Voet D, Voet J, Pratt C (2016). Fondamentaux de la biochimie . Hoboken, New Jersey : John Wiley & Sons, Inc. p. 336.ISBN _ 978-1-118-91840-1.
- ^ Chapman-Smith A, Cronan JE (1999). “La biotinylation enzymatique des protéines : une modification post-traductionnelle d’une spécificité exceptionnelle”. Tendances Biochem. Sci . 24 (9) : 359–363. doi : 10.1016/s0968-0004(99)01438-3 . PMID 10470036 .
- ^ Fisher Z, Hernandez Prada JA, Tu C, Duda D, Yoshioka C, An H, Govindasamy L, Silverman DN, McKenna R (février 2005). “Caractérisation structurelle et cinétique de l’histidine de site actif en tant que navette de protons en catalyse par l’anhydrase carbonique humaine II”. Biochimie . 44 (4): 1097-115. doi : 10.1021/bi0480279 . PMID 15667203 .
- ^ un b Wagner AL (1975). Vitamines et coenzymes . Krieger Pub Co.ISBN 0-88275-258-8.
- ^ “BRENDA Le système d’information complet sur les enzymes” . Technische Universität Braunschweig . Récupéré le 23 février 2015 .
- ^ Törnroth-Horsefield S, Neutze R (décembre 2008). “Ouverture et fermeture de la porte des métabolites” . Actes de l’Académie nationale des sciences des États-Unis d’Amérique . 105 (50): 19565–6. Bibcode : 2008PNAS..10519565T . doi : 10.1073/pnas.0810654106 . PMC 2604989 . PMID 19073922 .
- ^ McArdle WD, Katch F, Katch VL (2006). “Chapitre 9: Le système pulmonaire et l’exercice” . Essentials of Exercise Physiology (3e éd.). Baltimore, Maryland : Lippincott Williams & Wilkins. pp. 312–3. ISBN 978-0781749916.
- ^ Ferguson SJ, Nicholls D, Ferguson S (2002). Bioénergétique 3 (3e éd.). San Diego : Académique. ISBN 0-12-518121-3.
- ^ Bisswanger H (2017). Cinétique enzymatique : principes et méthodes (troisième éd. élargie et améliorée). Weinheim, Allemagne. ISBN 9783527806461. OCLC 992976641 .
- ^ Michaelis L, Menten M (1913). “Die Kinetik der Invertinwirkung” [La cinétique de l’action invertase]. Biochimie. Z. (en allemand). 49 : 333–369. ; Michaelis L, Menten ML, Johnson KA, Goody RS (2011). “La constante Michaelis originale: traduction de l’article Michaelis-Menten de 1913” . Biochimie . 50 (39): 8264–9. doi : 10.1021/bi201284u . PMC 3381512 . PMID 21888353 .
- ^ Briggs GE, Haldane JB (1925). “Une note sur la cinétique de l’action enzymatique” . Le Journal biochimique . 19 (2): 338–9. doi : 10.1042/bj0190338 . PMC 1259181 . PMID 16743508 .
- ^ Bar-Even A, Noor E, Savir Y, Liebermeister W, Davidi D, Tawfik DS, Milo R (2011). “L’enzyme modérément efficace: tendances évolutives et physico-chimiques façonnant les paramètres enzymatiques”. Biochimie . 50 (21): 4402–10. doi : 10.1021/bi2002289 . PMID 21506553 .
- ^ Ellis RJ (octobre 2001). “L’encombrement macromoléculaire: évident mais sous-estimé”. Tendances en sciences biochimiques . 26 (10): 597–604. doi : 10.1016/S0968-0004(01)01938-7 . PMID 11590012 .
- ^ Kopelman R (septembre 1988). “Cinétique de réaction fractale”. Sciences . 241 (4873): 1620–26. Bibcode : 1988Sci…241.1620K . doi : 10.1126/science.241.4873.1620 . PMID 17820893 . S2CID 23465446 .
- ^ un bcd Cornish – Bowden A (2004). Principes fondamentaux de la cinétique enzymatique (3 éd.). Londres : Portland Press. ISBN 1-85578-158-1.
- ^ Prix NC (1979). “Qu’entend-on par ‘inhibition compétitive’?”. Tendances en sciences biochimiques . 4 (11) : N272–N273. doi : 10.1016/0968-0004(79)90205-6 .
- ^ Goodsell DS (1er août 1999). « La perspective moléculaire : le méthotrexate » . L’oncologue . 4 (4): 340–341. doi : 10.1634/theoncologist.4-4-340 . ISSN 1083-7159 . PMID 10476546 .
- ^ Wu P, Clausen MH, Nielsen TE (décembre 2015). “Inhibiteurs allostériques de petites molécules de kinase” (PDF) . Pharmacologie & Thérapeutique . 156 : 59–68. doi : 10.1016/j.pharmthera.2015.10.002 . PMID 26478442 .
- ^ Cornish-Bowden A (juillet 1986). “Pourquoi l’inhibition non compétitive est-elle si rare? Une explication possible, avec des implications pour la conception de médicaments et de pesticides”. Lettres FEBS . 203 (1): 3–6. doi : 10.1016/0014-5793(86)81424-7 . PMID 3720956 . S2CID 45356060 .
- ^ Strelow JM (1er janvier 2017). “Une perspective sur la cinétique de l’inhibition covalente et irréversible” . Découverte SLAS . 22 (1): 3–20. doi : 10.1177/1087057116671509 . ISSN 2472-5552 . PMID 27703080 .
- ^ Fisher JF, Meroueh SO, Mobashery S (février 2005). “Résistance bactérienne aux Antibiotiques bêta-lactamines: opportunisme convaincant, opportunité convaincante”. Examens chimiques . 105 (2): 395–424. doi : 10.1021/cr030102i . PMID 15700950 .
- ^ un b Johnson DS, Weerapana E, Cravatt BF (juin 2010). “Stratégies pour découvrir et éliminer les risques d’inhibiteurs d’enzymes covalents et irréversibles” . Chimie médicinale du futur . 2 (6): 949–64. doi : 10.4155/fmc.10.21 . PMC 2904065 . PMID 20640225 .
- ^ Endo A (1er novembre 1992). “La découverte et le développement des inhibiteurs de la HMG-CoA réductase” . J. Lipid Res . 33 (11): 1569–82. doi : 10.1016/S0022-2275(20)41379-3 . PMID 1464741 .
- ^ Wlodawer A, Vondrasek J (1998). “Inhibiteurs de la protéase du VIH-1: un succès majeur de la conception de médicaments assistée par structure”. Revue annuelle de la biophysique et de la structure biomoléculaire . 27 : 249–84. doi : 10.1146/annurev.biophys.27.1.249 . PMID 9646869 . S2CID 10205781 .
- ^ Yoshikawa S, Caughey WS (mai 1990). “Preuve infrarouge de la liaison du cyanure aux sites de fer et de cuivre dans la cytochrome c oxydase cardiaque bovine. Implications concernant la réduction de l’oxygène” . Le Journal de chimie Biologique . 265 (14): 7945–58. doi : 10.1016/S0021-9258(19)39023-4 . PMID 2159465 .
- ^ Jain JL (mai 1999). Fondamentaux de la biochimie . New Delhi: S. Chand and Co. ISBN 8121903432. OCLC 818809626 .
- ^ Hunter T (janvier 1995). “Protéine kinases et phosphatases : le yin et le yang de la phosphorylation et de la signalisation des protéines” . Cellule . 80 (2): 225–236. doi : 10.1016/0092-8674(95)90405-0 . PMID 7834742 . S2CID 13999125 .
- ^ Berg JS, Powell BC, Cheney RE (avril 2001). “Un recensement millénaire de la myosine” . Biologie Moléculaire de la Cellule . 12 (4): 780–94. doi : 10.1091/mbc.12.4.780 . PMC 32266 . PMID 11294886 .
- ^ Meighen EA (mars 1991). “Biologie moléculaire de la bioluminescence bactérienne” . Examens microbiologiques . 55 (1): 123–142. doi : 10.1128/MMBR.55.1.123-142.1991 . PMC 372803 . PMID 2030669 .
- ^ De Clercq E (2002). “Faits saillants dans le développement de nouveaux agents antiviraux”. Mini Rev Med Chem . 2 (2): 163–75. doi : 10.2174/1389557024605474 . PMID 12370077 .
- ^ Mackie RI, White BA (octobre 1990). “Progrès récents dans l’écologie et le métabolisme microbiens du rumen : impact potentiel sur la production de nutriments” . Journal des sciences laitières . 73 (10): 2971–95. doi : 10.3168/jds.S0022-0302(90)78986-2 . PMID 2178174 .
- ^ Rouzer CA, Marnett LJ (2009). « Cyclooxygénases : aperçus structurels et fonctionnels » . J. Lipid Res . 50 Supplément : S29–34. doi : 10.1194/jlr.R800042-JLR200 . PMC 2674713 . PMID 18952571 .
- ^ un bcd Suzuki H (2015). “Chapitre 8: Contrôle de l’activité enzymatique”. Comment fonctionnent les enzymes : de la structure à la fonction . Boca Raton, Floride : CRC Press. p. 141–69. ISBN 978-981-4463-92-8.
- ^ Doble BW, Woodgett JR (avril 2003). « GSK-3 : trucs du métier pour une kinase multi-tâches » . Journal des sciences cellulaires . 116 (Pt 7): 1175–86. doi : 10.1242/jcs.00384 . PMC 3006448 . PMID 12615961 .
- ^ Bennett PM, Chopra I (1993). “Base moléculaire de l’induction de bêta-lactamase chez les bactéries” . Antimicrobien. Agents Chemother . 37 (2) : 153–8. doi : 10.1128/aac.37.2.153 . PMC 187630 . PMID 8452343 .
- ^ Skett P, Gibson GG (2001). “Chapitre 3: Induction et inhibition du métabolisme des médicaments”. Introduction au métabolisme des médicaments (3 éd.). Cheltenham, Royaume-Uni : Nelson Thornes Publishers. p. 87–118. ISBN 978-0748760114.
- ^ Faergeman NJ, Knudsen J (avril 1997). “Rôle des esters d’acyl-CoA gras à longue chaîne dans la régulation du métabolisme et dans la signalisation cellulaire” . Le Journal biochimique . 323 (Pt 1): 1–12. doi : 10.1042/bj3230001 . PMC 1218279 . PMID 9173866 .
- ^ Suzuki H (2015). “Chapitre 4: Effet du pH, de la température et de la haute pression sur l’activité enzymatique”. Comment fonctionnent les enzymes : de la structure à la fonction . Boca Raton, Floride : CRC Press. p. 53–74. ISBN 978-981-4463-92-8.
- ^ Noree C, Sato BK, Broyer RM, Wilhelm JE (août 2010). “Identification de nouvelles protéines formant des filaments chez Saccharomyces cerevisiae et Drosophila melanogaster” . Le Journal de biologie cellulaire . 190 (4): 541–51. doi : 10.1083/jcb.201003001 . PMC 2928026 . PMID 20713603 .
- ^ Aughey GN, Liu JL (2015). “Régulation métabolique via la filamentation enzymatique” . Revues critiques en biochimie et biologie moléculaire . 51 (4): 282-93. doi : 10.3109/10409238.2016.1172555 . PMC 4915340 . PMID 27098510 .
- ^ Kamata K, Mitsuya M, Nishimura T, Eiki J, Nagata Y (mars 2004). “Base structurelle pour la régulation allostérique de l’enzyme allostérique monomère glucokinase humaine” . Structure . 12 (3): 429-438. doi : 10.1016/j.str.2004.02.005 . PMID 15016359 .
- ^ Froguel P, Zouali H, Vionnet N, Velho G, Vaxillaire M, Sun F, Lesage S, Stoffel M, Takeda J, Passa P (mars 1993). “Hyperglycémie familiale due à des mutations de la glucokinase. Définition d’un sous-type de diabète sucré”. Le New England Journal of Medicine . 328 (10): 697–702. doi : 10.1056/NEJM199303113281005 . PMID 8433729 .
- ^ Okada S, O’Brien JS (août 1969). “Maladie de Tay-Sachs: absence généralisée d’un composant bêta-DN-acétylhexosaminidase”. Sciences . 165 (3894): 698–700. Bibcode : 1969Sci…165..698O . doi : 10.1126/science.165.3894.698 . PMID 5793973 . S2CID 8473726 .
- ^ “En savoir plus sur la maladie de Tay-Sachs” . Institut national américain de recherche sur le génome humain . Récupéré le 1er mars 2015 .
- ^ Erlandsen H, Stevens RC (octobre 1999). “La base structurelle de la phénylcétonurie”. Génétique moléculaire et métabolisme . 68 (2): 103–25. doi : 10.1006/mgme.1999.2922 . PMID 10527663 .
- ^ Flatmark T, Stevens RC (août 1999). “Aperçu structurel des hydroxylases d’acides aminés aromatiques et de leurs formes mutantes liées à la maladie”. Examens chimiques . 99 (8): 2137–2160. doi : 10.1021/cr980450y . PMID 11849022 .
- ^ “Phénylcétonurie” . Gènes et maladies [Internet] . Bethesda (MD) : Centre national d’information sur la biotechnologie (États-Unis). 1998–2015.
- ^ “Déficit en pseudocholinestérase” . Bibliothèque nationale de médecine des États-Unis . Récupéré le 5 septembre 2013 .
- ^ Fieker A, Philpott J, Armand M (2011). “Thérapie de remplacement d’enzymes pour l’insuffisance pancréatique : présent et futur” . Gastroentérologie Clinique et Expérimentale . 4 : 55–73. doi : 10.2147/CEG.S17634 . PMC 3132852 . PMID 21753892 .
- ^ Misselwitz B, Pohl D, Frühauf H, Fried M, Vavricka SR, Fox M (juin 2013). « Malabsorption et intolérance au lactose : pathogenèse, diagnostic et traitement » . Journal européen uni de gastroentérologie . 1 (3): 151–9. doi : 10.1177/2050640613484463 . PMC 4040760 . PMID 24917953 .
- ^ Cleaver JE (mai 1968). “Réplication de réparation défectueuse de l’ADN dans xeroderma pigmentosum”. Nature . 218 (5142): 652–6. Bibcode : 1968Natur.218..652C . doi : 10.1038/218652a0 . PMID 5655953 . S2CID 4171859 .
- ^ James WD, Elston D, Berger TG (2011). Maladies d’Andrews de la peau : dermatologie clinique (11e éd.). Londres : Saunders/ Elsevier. p. 567.ISBN _ 978-1437703146.
- ^ Murzin, AG (1993). “Les protéines homologues peuvent-elles développer différentes activités enzymatiques ?”. Tendances en sciences biochimiques . 18 (11): 403–405. doi : 10.1016/0968-0004(93)90132-7 . ISSN 0968-0004 . PMID 8291080 .
- ^ Ochoa D, Bradley D, Beltrao P (février 2018). “Evolution, dynamique et dysrégulation de la signalisation kinase”. Opinion actuelle en biologie structurale . 48 : 133–140. doi : 10.1016/j.sbi.2017.12.008 . PMID 29316484 .
- ^ Renugopalakrishnan V, Garduño-Juárez R, Narasimhan G, Verma CS, Wei X, Li P (novembre 2005). “Conception rationnelle de protéines thermiquement stables: pertinence pour la bionanotechnologie”. Journal des nanosciences et nanotechnologies . 5 (11): 1759-1767. doi : 10.1166/jnn.2005.441 . PMID 16433409 .
- ^ Hult K, Berglund P (août 2003). “Enzymes conçues pour une synthèse organique améliorée”. Opinion actuelle en biotechnologie . 14 (4): 395–400. doi : 10.1016/S0958-1669(03)00095-8 . PMID 12943848 .
- ^ Jiang L, Althoff EA, Clemente FR, Doyle L, Röthlisberger D, Zanghellini A, Gallaher JL, Betker JL, Tanaka F, Barbas CF, Hilvert D, Houk KN, Stoddard BL, Baker D (mars 2008). “Conception informatique de novo d’enzymes rétro-aldol” . Sciences . 319 (5868): 1387–91. Bib code : 2008Sci …319.1387J . doi : 10.1126/science.1152692 . PMC 3431203 . PMID 18323453 .
- ^ un b Sun Y, Cheng J (mai 2002). “Hydrolyse des matériaux lignocellulosiques pour la production d’éthanol : une revue”. Technologie des bioressources . 83 (1): 1–11. doi : 10.1016/S0960-8524(01)00212-7 . PMID 12058826 .
- ^ un b Kirk O, Borchert TV, Fuglsang CC (août 2002). “Applications d’enzymes industrielles”. Opinion actuelle en biotechnologie . 13 (4): 345–351. doi : 10.1016/S0958-1669(02)00328-2 . PMID 12323357 .
- ^ un bc Briggs DE (1998). Malts and Malting (1ère éd.). Londres : Blackie Academic. ISBN 978-0412298004.
- ^ Dulieu C, Moll M, Boudrant J, Poncelet D (2000). “Performances améliorées et contrôle de la fermentation de la bière à l’aide d’alpha-acétolactate décarboxylase encapsulée et modélisation”. Progrès de la biotechnologie . 16 (6): 958–65. doi : 10.1021/bp000128k . PMID 11101321 . S2CID 25674881 .
- ^ Tarté R (2008). Ingrédients des produits carnés Propriétés, fonctionnalités et applications . New York : Springer. p. 177. ISBN 978-0-387-71327-4.
- ^ “Chymosine – Base de données OGM” . Boussole OGM . Union européenne. 10 juillet 2010. Archivé de l’original le 26 mars 2015 . Récupéré le 1er mars 2015 .
- ^ Molimard P, Spinnler HE (février 1996). “Revue : Composés impliqués dans la saveur des fromages affinés à la moisissure en surface : origines et propriétés” . Journal des sciences laitières . 79 (2): 169-184. doi : 10.3168/jds.S0022-0302(96)76348-8 .
- ^ Guzmán-Maldonado H, Paredes-López O (septembre 1995). “Enzymes amylolytiques et produits dérivés de l’amidon: une revue”. Revues critiques en science alimentaire et nutrition . 35 (5): 373–403. doi : 10.1080/10408399509527706 . PMID 8573280 .
- ^ un b “Protéase – Base de données d’OGM” . Boussole OGM . Union européenne. 10 juillet 2010. Archivé de l’original le 24 février 2015 . Récupéré le 28 février 2015 .
- ^ Alkorta I, Garbisu C, Llama MJ, Serra JL (janvier 1998). “Applications industrielles des enzymes pectiques : une revue”. Biochimie des procédés . 33 (1): 21–28. doi : 10.1016/S0032-9592(97)00046-0 .
- ^ Bajpai P (mars 1999). “Application des enzymes dans l’industrie des pâtes et papiers”. Progrès de la biotechnologie . 15 (2): 147-157. doi : 10.1021/bp990013k . PMID 10194388 . S2CID 26080240 .
- ^ Begley CG, Paragina S, Sporn A (mars 1990). “Une analyse des nettoyants enzymatiques pour lentilles de contact”. Journal de l’Association optométrique américaine . 61 (3) : 190–4. PMID 2186082 .
- ^ Farris PL (2009). “Croissance économique et organisation de l’industrie américaine de l’amidon”. Dans BeMiller JN, Whistler RL (eds.). Chimie et technologie de l’amidon (3e éd.). Londres : Académique. ISBN 9780080926551.
Lectures complémentaires
Général
Étymologie et histoire
|
Structure et mécanisme enzymatique
Cinétique et inhibition
|