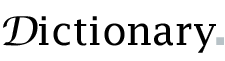Biosynthèse des protéines
La biosynthèse des protéines (ou synthèse des protéines ) est un processus biologique central, se produisant à l’intérieur des cellules , équilibrant la perte de protéines cellulaires (via la dégradation ou l’exportation ) par la production de nouvelles protéines. Les protéines remplissent un certain nombre de fonctions essentielles comme les enzymes , les protéines structurelles ou les hormones . La synthèse des protéines est un processus très similaire pour les procaryotes et les eucaryotes , mais il existe des différences distinctes. [1]
 Biosynthèse des protéines à partir de la transcription et des modifications post-transcriptionnelles dans le noyau. Ensuite, l’ARNm mature est exporté vers le cytoplasme où il est traduit. La Chaîne polypeptidique se replie ensuite et est modifiée après la traduction.
Biosynthèse des protéines à partir de la transcription et des modifications post-transcriptionnelles dans le noyau. Ensuite, l’ARNm mature est exporté vers le cytoplasme où il est traduit. La Chaîne polypeptidique se replie ensuite et est modifiée après la traduction.
La synthèse des protéines peut être globalement divisée en deux phases : la transcription et la traduction . Au cours de la transcription, une section d’ ADN codant pour une protéine, appelée gène , est convertie en une molécule matrice appelée ARN messager (ARNm). Cette conversion est effectuée par des enzymes, appelées ARN polymérases , dans le noyau de la cellule . [2] Chez les eucaryotes, cet ARNm est initialement produit sous une forme prématurée ( pré-ARNm ) qui subit des modifications post-transcriptionnelles pour produire des ARNm matures . L’ARNm mature est exporté du noyau cellulaire viapores nucléaires vers le cytoplasme de la cellule pour que la traduction se produise. Lors de la traduction, l’ARNm est lu par des ribosomes qui utilisent la séquence nucléotidique de l’ARNm pour déterminer la séquence d’ acides aminés . Les ribosomes catalysent la formation de liaisons peptidiques covalentes entre les acides aminés codés pour former une Chaîne polypeptidique .
Après la traduction, la Chaîne polypeptidique doit se replier pour former une protéine fonctionnelle ; par exemple, pour fonctionner comme une enzyme, la Chaîne polypeptidique doit se replier correctement pour produire un site actif fonctionnel . Afin d’adopter une forme tridimensionnelle (3D) fonctionnelle, la Chaîne polypeptidique doit d’abord former une série de structures sous-jacentes plus petites appelées structures secondaires . La Chaîne polypeptidique de ces structures secondaires se replie ensuite pour produire la structure tertiaire 3D globale . Une fois correctement repliée, la protéine peut subir une maturation supplémentaire à travers différentes modifications post-traductionnelles. Les modifications post-traductionnelles peuvent modifier la capacité de la protéine à fonctionner, son emplacement dans la cellule (par exemple, le cytoplasme ou le noyau) et la capacité de la protéine à interagir avec d’autres protéines . [3]
La biosynthèse des protéines joue un rôle clé dans la maladie, car les changements et les erreurs dans ce processus, par le biais de mutations sous-jacentes de l’ADN ou d’un mauvais repliement des protéines, sont souvent les causes sous-jacentes d’une maladie. Les mutations de l’ADN modifient la séquence d’ARNm ultérieure, qui modifie ensuite la séquence d’acides aminés codée par l’ARNm. Les mutations peuvent raccourcir la Chaîne polypeptidique en générant une séquence d’arrêt qui provoque l’arrêt précoce de la traduction. En variante, une mutation dans la séquence d’ARNm modifie l’acide aminé spécifique codé à cette position dans la Chaîne polypeptidique. Ce changement d’acide aminé peut avoir un impact sur la capacité de la protéine à fonctionner ou à se replier correctement. [4]Les protéines mal repliées sont souvent impliquées dans la maladie, car les protéines mal repliées ont tendance à s’agglutiner pour former des amas protéiques denses . Ces amas sont liés à un éventail de maladies, souvent neurologiques , dont la maladie d’Alzheimer et la maladie de Parkinson . [5]
Transcription
La transcription se produit dans le noyau en utilisant l’ADN comme matrice pour produire de l’ARNm. Chez les eucaryotes, cette molécule d’ARNm est connue sous le nom de pré-ARNm car elle subit des modifications post-transcriptionnelles dans le noyau pour produire une molécule d’ARNm mature. Cependant, chez les procaryotes, des modifications post-transcriptionnelles ne sont pas nécessaires, de sorte que la molécule d’ARNm mature est immédiatement produite par transcription. [1]

 Illustrer la structure d’un nucléotide avec les 5 carbones marqués démontrant la nature 5 ‘du groupe phosphate et la nature 3’ du groupe Hydroxyle nécessaires pour former les Liaisons phosphodiester conjonctives
Illustrer la structure d’un nucléotide avec les 5 carbones marqués démontrant la nature 5 ‘du groupe phosphate et la nature 3’ du groupe Hydroxyle nécessaires pour former les Liaisons phosphodiester conjonctives 
 Illustre la directionnalité intrinsèque de la molécule d’ADN avec le brin codant allant de 5′ à 3′ et le brin matrice complémentaire allant de 3′ à 5′
Illustre la directionnalité intrinsèque de la molécule d’ADN avec le brin codant allant de 5′ à 3′ et le brin matrice complémentaire allant de 3′ à 5′
Initialement, une enzyme connue sous le nom d’ hélicase agit sur la molécule d’ADN. L’ADN a une structure en double hélice antiparallèle composée de deux brins polynucléotidiques complémentaires , maintenus ensemble par des liaisons hydrogène entre les paires de bases. L’hélicase perturbe les liaisons hydrogène provoquant le déroulement d’une région d’ADN – correspondant à un gène -, séparant les deux brins d’ADN et exposant une série de bases. Bien que l’ADN soit une molécule double brin, un seul des brins agit comme matrice pour la synthèse du pré-ARNm – ce brin est connu sous le nom de brin matrice. L’autre brin d’ADN (qui est complémentaire du brin matrice) est appelé brin codant. [6]
L’ADN et l’ARN ont tous deux une directionnalité intrinsèque , ce qui signifie qu’il existe deux extrémités distinctes de la molécule. Cette propriété de directionnalité est due aux sous-unités nucléotidiques sous-jacentes asymétriques, avec un groupe phosphate d’un côté du sucre pentose et une base de l’autre. Les cinq carbones du sucre pentose sont numérotés de 1′ (où ‘ signifie premier) à 5’. Par conséquent, les Liaisons phosphodiester reliant les nucléotides sont formées en joignant le groupe Hydroxyle sur le carbone 3′ d’un nucléotide au groupe phosphate sur le carbone 5′ d’un autre nucléotide. Par conséquent, le brin codant de l’ADN s’étend dans une direction 5 ‘à 3′ et le brin d’ADN matrice complémentaire s’étend dans la direction opposée de 3′ à 5’. [1]
 Illustre la conversion du brin matrice d’ADN en molécule de pré-ARNm par l’ARN polymérase.
Illustre la conversion du brin matrice d’ADN en molécule de pré-ARNm par l’ARN polymérase.
L’enzyme ARN polymérase se lie au brin matrice exposé et se lit à partir du gène dans la direction 3′ vers 5′. Simultanément, l’ARN polymérase synthétise un seul brin de pré-ARNm dans le sens 5′ vers 3′ en catalysant la formation de Liaisons phosphodiester entre les nucléotides activés (libres dans le noyau) capables d’ appariement de bases complémentaires avec le brin matrice. . Derrière l’ARN polymérase en mouvement, les deux brins d’ADN se rejoignent, de sorte que seulement 12 paires de bases d’ADN sont exposées à la fois. [6]L’ARN polymérase construit la molécule de pré-ARNm à une vitesse de 20 nucléotides par seconde permettant la production de milliers de molécules de pré-ARNm à partir du même gène en une heure. Malgré le taux de synthèse rapide, l’enzyme ARN polymérase contient son propre mécanisme de relecture. Les mécanismes de relecture permettent à l’ARN polymérase d’éliminer les nucléotides incorrects (qui ne sont pas complémentaires du brin matrice d’ADN) de la molécule de pré-ARNm en croissance par une réaction d’excision. [1] Lorsque l’ARN polymérase atteint une séquence d’ADN spécifique qui termine la transcription, l’ARN polymérase se détache et la synthèse du pré-ARNm est terminée. [6]
La molécule de pré-ARNm synthétisée est complémentaire du brin d’ADN matrice et partage la même séquence de nucléotides que le brin d’ADN codant. Cependant, il existe une différence cruciale dans la composition en nucléotides des molécules d’ADN et d’ARNm. L’ADN est composé des bases – guanine , cytosine , adénine et thymine (G, C, A et T) – L’ARN est également composé de quatre bases – guanine, cytosine, adénine et uracile . Dans les molécules d’ARN, la thymine de base de l’ADN est remplacée par l’uracile qui est capable de s’apparier avec l’adénine. Par conséquent, dans la molécule de pré-ARNm, toutes les bases complémentaires qui seraient la thymine dans le brin d’ADN codant sont remplacées par l’uracile. [7]
Modifications post-transcriptionnelles

 Décrit le processus de modification post-transcriptionnelle du pré-ARNm par coiffage, polyadénylation et épissage pour produire une molécule d’ARNm mature prête à être exportée du noyau.
Décrit le processus de modification post-transcriptionnelle du pré-ARNm par coiffage, polyadénylation et épissage pour produire une molécule d’ARNm mature prête à être exportée du noyau.
Une fois la transcription terminée, la molécule de pré-ARNm subit des modifications post-transcriptionnelles pour produire une molécule d’ARNm mature.
Il y a 3 étapes clés dans les modifications post-transcriptionnelles :
- Ajout d’une coiffe 5′ à l’extrémité 5′ de la molécule de pré-ARNm
- L’ajout d’une queue poly (A) 3 ‘ est ajouté à la molécule de pré-ARNm de l’extrémité 3’
- Élimination des introns par épissage d’ARN
La coiffe 5′ est ajoutée à l’extrémité 5′ de la molécule de pré-ARNm et est composée d’un nucléotide guanine modifié par méthylation . Le but de la coiffe 5 ‘est d’empêcher la décomposition des molécules d’ARNm matures avant la traduction, la coiffe facilite également la liaison du ribosome à l’ARNm pour démarrer la traduction [8] et permet de différencier l’ARNm des autres ARN dans la cellule. [1] En revanche, la queue 3′ Poly(A) est ajoutée à l’extrémité 3′ de la molécule d’ARNm et est composée de 100 à 200 bases d’adénine. [8] Ces modifications distinctes de l’ARNm permettent à la cellule de détecter que le message d’ARNm complet est intact si à la fois la coiffe 5′ et la queue 3′ sont présentes. [1]
Cette molécule de pré-ARNm modifiée subit ensuite le processus d’épissage d’ARN. Les gènes sont composés d’une série d’introns et d’ exons , les introns sont des séquences de nucléotides qui ne codent pas une protéine tandis que les exons sont des séquences de nucléotides qui codent directement une protéine. Les introns et les exons sont présents à la fois dans la séquence d’ADN sous-jacente et dans la molécule de pré-ARNm. Par conséquent, afin de produire une molécule d’ARNm mature codant pour une protéine, un épissage doit avoir lieu. [6] Au cours de l’épissage, les introns intermédiaires sont retirés de la molécule de pré-ARNm par un complexe multiprotéique connu sous le nom de spliceosome (composé de plus de 150 protéines et ARN). [9] Cette molécule d’ARNm mature est ensuite exportée dans le cytoplasme à travers les pores nucléaires de l’enveloppe du noyau.
Traduction

 Illustre le processus de traduction montrant le cycle d’appariement codon-anti-codon de l’ARNt et l’incorporation d’acides aminés dans la Chaîne polypeptidique en croissance par le ribosome.
Illustre le processus de traduction montrant le cycle d’appariement codon-anti-codon de l’ARNt et l’incorporation d’acides aminés dans la Chaîne polypeptidique en croissance par le ribosome. 
 Un ribosome sur un brin d’ARNm avec des ARNt arrivant, effectuant un appariement codon-anti-codon base, délivrant leur acide aminé à la Chaîne polypeptidique en croissance et partant. Démontre l’action du ribosome en tant que machine biologique qui fonctionne à l’échelle nanométrique pour effectuer la traduction. Le ribosome se déplace le long de la molécule d’ARNm mature incorporant l’ARNt et produisant une Chaîne polypeptidique.
Un ribosome sur un brin d’ARNm avec des ARNt arrivant, effectuant un appariement codon-anti-codon base, délivrant leur acide aminé à la Chaîne polypeptidique en croissance et partant. Démontre l’action du ribosome en tant que machine biologique qui fonctionne à l’échelle nanométrique pour effectuer la traduction. Le ribosome se déplace le long de la molécule d’ARNm mature incorporant l’ARNt et produisant une Chaîne polypeptidique.
Au cours de la traduction, les ribosomes synthétisent des chaînes polypeptidiques à partir de molécules matrices d’ARNm. Chez les eucaryotes, la traduction se produit dans le cytoplasme de la cellule, où les ribosomes flottent librement ou sont attachés au réticulum endoplasmique . Chez les procaryotes, dépourvus de noyau, les processus de transcription et de traduction se produisent dans le cytoplasme. [dix]
Les ribosomes sont des machines moléculaires complexes , constituées d’un mélange de protéine et d’ARN ribosomal , disposées en deux sous-unités (une grande et une petite sous-unité), qui entourent la molécule d’ARNm. Le ribosome lit la molécule d’ARNm dans une direction 5′-3′ et l’utilise comme matrice pour déterminer l’ordre des acides aminés dans la Chaîne polypeptidique. [11] Afin de traduire la molécule d’ARNm, le ribosome utilise de petites molécules, appelées ARN de transfert(ARNt), pour délivrer les acides aminés corrects au ribosome. Chaque ARNt est composé de 70 à 80 nucléotides et adopte une structure caractéristique en trèfle en raison de la formation de liaisons hydrogène entre les nucléotides au sein de la molécule. Il existe environ 60 types différents d’ARNt, chaque ARNt se lie à une séquence spécifique de trois nucléotides (appelée codon ) dans la molécule d’ARNm et délivre un acide aminé spécifique. [12]
Le ribosome se fixe initialement à l’ARNm au niveau du codon de départ (AUG) et commence à traduire la molécule. La séquence nucléotidique de l’ARNm est lue en triplets- trois nucléotides adjacents dans la molécule d’ARNm correspondent à un seul codon. Chaque ARNt a une séquence exposée de trois nucléotides, connus sous le nom d’anticodon, dont la séquence est complémentaire d’un codon spécifique qui peut être présent dans l’ARNm. Par exemple, le premier codon rencontré est le codon start composé des nucléotides AUG. L’ARNt correct avec l’anticodon (séquence complémentaire de 3 nucléotides UAC) se lie à l’ARNm à l’aide du ribosome. Cet ARNt délivre le bon acide aminé correspondant au codon de l’ARNm, dans le cas du codon de départ, il s’agit de l’acide aminé méthionine. Le codon suivant (adjacent au codon de départ) est alors lié par l’ARNt correct avec un anticodon complémentaire, délivrant l’acide aminé suivant au ribosome. Le ribosome utilise alors sa peptidyl transféraseactivité enzymatique pour catalyser la formation de la liaison peptidique covalente entre les deux acides aminés adjacents. [6]
Le ribosome se déplace ensuite le long de la molécule d’ARNm jusqu’au troisième codon. Le ribosome libère alors la première molécule d’ARNt, car seules deux molécules d’ARNt peuvent être réunies par un seul ribosome à la fois. L’ARNt complémentaire suivant avec l’anticodon correct complémentaire du troisième codon est sélectionné, délivrant l’acide aminé suivant au ribosome qui est joint de manière covalente à la Chaîne polypeptidique en croissance. Ce processus se poursuit avec le ribosome se déplaçant le long de la molécule d’ARNm ajoutant jusqu’à 15 acides aminés par seconde à la Chaîne polypeptidique. Derrière le premier ribosome, jusqu’à 50 ribosomes supplémentaires peuvent se lier à la molécule d’ARNm formant un polysome , ce qui permet la synthèse simultanée de plusieurs chaînes polypeptidiques identiques. [6]La terminaison de la Chaîne polypeptidique en croissance se produit lorsque le ribosome rencontre un codon d’arrêt (UAA, UAG ou UGA) dans la molécule d’ARNm. Lorsque cela se produit, aucun ARNt ne peut le reconnaître et un facteur de libération induit la libération de la Chaîne polypeptidique complète du ribosome. [12] Le Dr Har Gobind Khorana , un scientifique originaire d’Inde, a décodé les séquences d’ARN pour environ 20 acides aminés. [ la citation nécessaire ] Il a reçu le Prix Nobel en 1968, avec deux autres scientifiques, pour son travail.
Repliement des protéines

 Montre le processus de repliement d’une Chaîne polypeptidique depuis sa structure primaire initiale jusqu’à la structure quaternaire.
Montre le processus de repliement d’une Chaîne polypeptidique depuis sa structure primaire initiale jusqu’à la structure quaternaire.
Une fois la synthèse de la Chaîne polypeptidique terminée, la Chaîne polypeptidique se replie pour adopter une structure spécifique qui permet à la protéine de remplir ses fonctions. La forme de base de la structure protéique est connue sous le nom de structure primaire , qui est simplement la Chaîne polypeptidique, c’est-à-dire une séquence d’acides aminés liés de manière covalente. La structure primaire d’une protéine est codée par un gène. Par conséquent, toute modification de la séquence du gène peut modifier la structure primaire de la protéine et tous les niveaux ultérieurs de la structure protéique, modifiant finalement la structure et la fonction globales.
La structure primaire d’une protéine (la Chaîne polypeptidique) peut alors se replier ou s’enrouler pour former la structure secondaire de la protéine. Les types les plus courants de structure secondaire sont connus sous le nom d’ hélice alpha ou de feuillet bêta , ce sont de petites structures produites par des liaisons hydrogène se formant au sein de la Chaîne polypeptidique. Cette structure secondaire se replie ensuite pour produire la structure tertiaire de la protéine. La structure tertiaire est la structure 3D globale des protéines qui est constituée de différentes structures secondaires se repliant ensemble. Dans la structure tertiaire, les caractéristiques clés de la protéine, par exemple le site actif, sont repliées et formées, permettant à la protéine de fonctionner. Enfin, certaines protéines peuvent adopter une structure quaternaire complexe. La plupart des protéines sont constituées d’une seule Chaîne polypeptidique, cependant, certaines protéines sont composées de plusieurs chaînes polypeptidiques (appelées sous-unités) qui se replient et interagissent pour former la structure quaternaire. Par conséquent, la protéine globale est un complexe multi-sous-unités composé de plusieurs sous-unités de chaînes polypeptidiques repliées, par exemple l’Hémoglobine . [13]
Modifications post-traductionnelles
Lorsque la protéine se replie dans l’état 3D mature et fonctionnel est terminée, ce n’est pas nécessairement la fin de la voie de maturation de la protéine. Une protéine repliée peut encore subir un traitement supplémentaire par des modifications post-traductionnelles. Il existe plus de 200 types connus de modifications post-traductionnelles, ces modifications peuvent altérer l’activité protéique, la capacité de la protéine à interagir avec d’autres protéines et où la protéine se trouve dans la cellule, par exemple dans le noyau ou le cytoplasme cellulaire. [14] Grâce à des modifications post-traductionnelles, la diversité des protéines codées par le génome est élargie de 2 à 3 ordres de grandeur . [15]
Il existe quatre classes clés de modification post-traductionnelle : [3]
- Clivage
- Ajout de groupes chimiques
- Ajout de molécules complexes
- Formation de liaisons intramoléculaires
Clivage
Polypeptide chain, one chain is intact with three arrows indicating sites of protease cleavage on the chain and intermolecular disulphide bonds. The second chain is in three pieces connected by disulphide bonds.” height=”147″ data-src=”//upload.wikimedia.org/wikipedia/commons/thumb/7/7d/Post-translational_modification_by_cleavage.png/400px-Post-translational_modification_by_cleavage.png” width=”400″> Montre une modification post-traductionnelle de la protéine par clivage par protéase, illustrant que les liaisons préexistantes sont conservées même si la Chaîne polypeptidique est clivée.
Le clivage des protéines est une modification post-traductionnelle irréversible réalisée par des enzymes appelées Protéases . Ces Protéases sont souvent très spécifiques et provoquent l’ hydrolyse d’un nombre limité de liaisons peptidiques au sein de la protéine cible. La protéine raccourcie résultante a une Chaîne polypeptidique altérée avec différents acides aminés au début et à la fin de la chaîne. Cette modification post-traductionnelle altère souvent la fonction des protéines, la protéine peut être inactivée ou activée par le clivage et peut présenter de nouvelles activités biologiques. [16]
Ajout de groupes chimiques

 Montre la modification post-traductionnelle de la protéine par méthylation, acétylation et phosphorylation
Montre la modification post-traductionnelle de la protéine par méthylation, acétylation et phosphorylation
Après traduction, de petits groupes chimiques peuvent être ajoutés aux acides aminés dans la structure protéique mature. [17] Des exemples de processus qui ajoutent des groupes chimiques à la protéine cible comprennent la méthylation, l’ acétylation et la phosphorylation .
La méthylation est l’addition réversible d’un groupe méthyle sur un acide aminé catalysée par des enzymes méthyltransférases . La méthylation se produit sur au moins 9 des 20 acides aminés courants, cependant, elle se produit principalement sur les acides aminés lysine et arginine . Un exemple d’une protéine couramment méthylée est une histone . Les histones sont des protéines présentes dans le noyau de la cellule. L’ADN est étroitement enroulé autour des histones et maintenu en place par d’autres protéines et des interactions entre les charges négatives de l’ADN et les charges positives de l’histone. Un schéma très spécifique de méthylation des acides aminéssur les protéines histones est utilisé pour déterminer quelles régions d’ADN sont étroitement enroulées et incapables d’être transcrites et quelles régions sont lâchement enroulées et capables d’être transcrites. [18]
La régulation basée sur les histones de la transcription de l’ADN est également modifiée par l’acétylation. L’acétylation est l’addition covalente réversible d’un groupe acétyle sur un acide aminé lysine par l’enzyme acétyltransférase . Le groupe acétyle est retiré d’une molécule donneuse connue sous le nom d’ acétyl coenzyme A et transféré sur la protéine cible. [19] Les histones subissent une acétylation sur leurs résidus de lysine par des enzymes connues sous le nom d’ histone acétyltransférase . L’effet de l’acétylation est d’affaiblir les interactions de charge entre l’histone et l’ADN, rendant ainsi plus de gènes dans l’ADN accessibles pour la transcription. [20]
La modification de groupe chimique post-traductionnelle finale et répandue est la phosphorylation. La phosphorylation est l’addition réversible et covalente d’un groupe phosphate à des acides aminés spécifiques ( sérine , thréonine et tyrosine ) au sein de la protéine. Le groupe phosphate est retiré de la molécule donneuse ATP par une protéine kinase et transféré sur le groupe Hydroxyle de l’acide aminé cible, ce qui produit de l’ adénosine diphosphate comme sous-produit. Ce processus peut être inversé et le groupe phosphate éliminé par l’enzyme protéine phosphatase. La phosphorylation peut créer un site de liaison sur la protéine phosphorylée qui lui permet d’interagir avec d’autres protéines et de générer de grands complexes multiprotéiques. Alternativement, la phosphorylation peut modifier le niveau d’activité protéique en modifiant la capacité de la protéine à se lier à son substrat. [1]
Ajout de molécules complexes
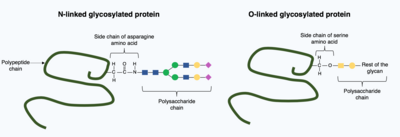
 Illustre la différence de structure entre la glycosylation N-liée et O-liée sur une Chaîne polypeptidique.
Illustre la différence de structure entre la glycosylation N-liée et O-liée sur une Chaîne polypeptidique.
Les modifications post-traductionnelles peuvent incorporer des molécules plus complexes et plus grandes dans la structure protéique repliée. Un exemple courant de ceci est la glycosylation , l’ajout d’une molécule de polysaccharide, qui est largement considérée comme la modification post-traductionnelle la plus courante. [15]
Dans la glycosylation, une molécule de polysaccharide (appelée glycane ) est ajoutée de manière covalente à la protéine cible par des enzymes Glycosyltransférases et modifiée par des glycosidases dans le réticulum endoplasmique et l’appareil de Golgi . La glycosylation peut jouer un rôle essentiel dans la détermination de la structure 3D finale et pliée de la protéine cible. Dans certains cas, la glycosylation est nécessaire pour un repliement correct. La glycosylation N-liée favorise le repliement des protéines en augmentant la solubilité et médie la liaison des protéines aux chaperons protéiques . Les chaperons sont des protéines responsables du repliement et du maintien de la structure d’autres protéines. [1]
Il existe en gros deux types de glycosylation, la glycosylation N-liée et la glycosylation O-liée . La glycosylation N-liée commence dans le réticulum endoplasmique avec l’ajout d’un glycane précurseur. Le glycane précurseur est modifié dans l’appareil de Golgi pour produire un glycane complexe lié de manière covalente à l’azote dans un acide aminé d’ asparagine . En revanche, la glycosylation liée à O est l’addition covalente séquentielle de sucres individuels sur l’oxygène dans les acides aminés sérine et thréonine dans la structure protéique mature. [1]
Formation de liaisons covalentes
Polypeptide chain and formation of a disulphide bond between two cysteine amino acids on different polypeptide chains, thereby joining the two chains.” height=”285″ data-src=”//upload.wikimedia.org/wikipedia/commons/thumb/f/f6/Formation_of_disulphide_covalent_bonds.png/400px-Formation_of_disulphide_covalent_bonds.png” width=”400″> Montre la formation de liaisons covalentes disulfure en tant que modification post-traductionnelle. Des liaisons disulfure peuvent se former soit au sein d’une seule Chaîne polypeptidique (à gauche), soit entre des chaînes polypeptidiques dans un complexe protéique multi-sous-unité (à droite).
De nombreuses protéines produites dans la cellule sont sécrétées à l’extérieur de la cellule pour fonctionner comme des protéines extracellulaires . Les protéines extracellulaires sont exposées à une grande variété de conditions. Afin de stabiliser la structure protéique 3D, des liaisons covalentes se forment soit au sein de la protéine, soit entre les différentes chaînes polypeptidiques de la structure quaternaire. Le type le plus répandu est une liaison disulfure (également connue sous le nom de pont disulfure). Une liaison disulfure est formée entre deux acides aminés cystéine en utilisant leurs groupes chimiques de chaîne latérale contenant un atome de soufre, ces groupes chimiques sont appelés groupes fonctionnels thiol . Les liaisons disulfure agissent pour stabiliser la structure préexistantede la protéine. Les liaisons disulfure sont formées dans une réaction d’oxydation entre deux groupes thiol et ont donc besoin d’un environnement oxydant pour réagir. En conséquence, des liaisons disulfure sont généralement formées dans l’environnement oxydant du réticulum endoplasmique catalysé par des enzymes appelées protéine disulfure isomérases. Les liaisons disulfure sont rarement formées dans le cytoplasme car il s’agit d’un environnement réducteur. [1]
Rôle de la synthèse des protéines dans la maladie
De nombreuses maladies sont causées par des mutations dans les gènes, en raison de la connexion directe entre la séquence nucléotidique de l’ADN et la séquence d’acides aminés de la protéine codée. Des modifications de la structure primaire de la protéine peuvent entraîner un mauvais repliement ou un dysfonctionnement de la protéine. Des mutations au sein d’un même gène ont été identifiées comme une cause de plusieurs maladies, y compris la drépanocytose , connue sous le nom de troubles monogéniques.
Drépanocytose

 Une comparaison entre un individu en bonne santé et une personne atteinte d’anémie falciforme illustrant les différentes formes de globules rouges et les différents flux sanguins dans les vaisseaux sanguins.
Une comparaison entre un individu en bonne santé et une personne atteinte d’anémie falciforme illustrant les différentes formes de globules rouges et les différents flux sanguins dans les vaisseaux sanguins.
La drépanocytose est un groupe de maladies causées par une mutation dans une sous-unité de l’Hémoglobine, une protéine présente dans les globules rouges responsable du transport de l’oxygène. La plus dangereuse des drépanocytoses est connue sous le nom d’anémie falciforme. L’anémie falciforme est la maladie monogénique homozygote récessive la plus courante , ce qui signifie que la personne atteinte doit être porteuse d’une mutation dans les deux copies du gène affecté (une héritée de chaque parent) pour souffrir de la maladie. L’Hémoglobine a une structure quaternaire complexe et est composée de quatre sous-unités polypeptidiques – deux sous-unités A et deux sous-unités B. [21]Les patients souffrant d’anémie falciforme ont une mutation faux-sens ou de substitution dans le gène codant pour la Chaîne polypeptidique de la sous-unité B de l’Hémoglobine. Une mutation faux-sens signifie que la mutation nucléotidique modifie le triplet de codons global de sorte qu’un acide aminé différent est associé au nouveau codon. Dans le cas de la drépanocytose, la mutation faux-sens la plus courante est une mutation d’un seul nucléotide de la thymine à l’adénine dans le gène de la sous-unité B de l’Hémoglobine. [22] Cela change le codon 6 du codage de l’acide glutamique d’acide aminé au codage de la valine. [21]
Ce changement dans la structure primaire de la Chaîne polypeptidique de la sous-unité B de l’Hémoglobine modifie la fonctionnalité du complexe multi-sous-unité de l’Hémoglobine dans des conditions de faible teneur en oxygène. Lorsque les globules rouges déchargent de l’oxygène dans les tissus du corps, la protéine d’Hémoglobine mutée commence à se coller pour former une structure semi-solide dans le globule rouge. Cela déforme la forme du globule rouge, ce qui lui donne la forme caractéristique de “faucille” et réduit la flexibilité des cellules. Ce globule rouge rigide et déformé peut s’accumuler dans les vaisseaux sanguins et créer un blocage. Le blocage empêche le flux sanguin vers les tissus et peut entraîner la mort des tissus, ce qui cause une grande douleur à l’individu. [23]
Cancer
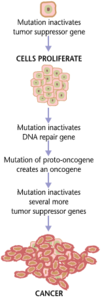
 Formation de gènes cancéreux due à un dysfonctionnement des gènes suppresseurs.
Formation de gènes cancéreux due à un dysfonctionnement des gènes suppresseurs.
Les cancers se forment à la suite de mutations génétiques ainsi que d’une mauvaise traduction des protéines. En plus des cellules cancéreuses proliférant anormalement, ils suppriment l’expression de gènes ou de protéines anti-apoptotique ou pro-apoptotique. La plupart des cellules cancéreuses voient une mutation dans la protéine de signalisation Ras, qui fonctionne comme un transducteur de signal marche/arrêt dans les cellules. Dans les cellules cancéreuses, la protéine RAS devient active de manière persistante, favorisant ainsi la prolifération de la cellule en l’absence de toute régulation. [24] De plus, la plupart des cellules cancéreuses portent deux copies mutantes du gène régulateur p53, qui agit comme un gardien pour les gènes endommagés et initie l’apoptose dans les cellules malignes. En son absence, la cellule ne peut pas initier l’apoptose ou signaler à d’autres cellules de la détruire. [25]
Au fur et à mesure que les cellules tumorales prolifèrent, soit elles restent confinées à une zone et sont dites bénignes, soit elles deviennent des cellules malignes qui migrent vers d’autres zones du corps. Souvent, ces cellules malignes sécrètent des Protéases qui brisent la matrice Extracellulaire des tissus. Cela permet alors au cancer d’entrer dans sa phase terminale appelée métastase, dans laquelle les cellules pénètrent dans la circulation sanguine ou le système lymphatique pour se rendre dans une nouvelle partie du corps. [24]
Voir également
- Dogme central de la biologie moléculaire
- Code génétique
- L’expression du gène
- Modification post-traductionnelle
- Repliement des protéines
Références
- ^ un bcdefghij Alberts B ( 2015 ) . _ _ _ Biologie moléculaire de la cellule (sixième éd.). Abingdon, Royaume-Uni : Garland Science, Taylor and Francis Group. ISBN 978-0815344643.
- ^ O’Connor C (2010). L’essentiel de la biologie cellulaire . Éducation de NPG : Cambridge, mA . Récupéré le 3 mars 2020 .
- ^ un b Wang YC, Peterson SE, Loring JF (février 2014). “Modifications post-traductionnelles des protéines et régulation de la pluripotence dans les cellules souches humaines” . Recherche cellulaire . 24 (2): 143–160. doi : 10.1038/cr.2013.151 . PMC 3915910 . PMID 24217768 .
- ^ Scheper GC, van der Knaap MS, Proud CG (septembre 2007). “La traduction importe: les défauts de synthèse des protéines dans les maladies héréditaires”. Revues naturelles. Génétique . 8 (9): 711–723. doi : 10.1038/nrg2142 . PMID 17680008 . S2CID 12153982 .
- ^ Berg JM, Tymoczko JL, Gatto Jr GJ, Stryer L (2015). Biochimie (huitième éd.). États-Unis : WH Freeman and Company. ISBN 9781464126109.
- ^ un bcdef Toole G , Toole S (2015). AQA biologie niveau A. Livre étudiant (deuxième éd.). Great Clarendon Street, Oxford, OX2 6DP, Royaume-Uni : Oxford University Press. ISBN 9780198351771.{{cite book}}: Maint CS1: emplacement ( lien )
- ^ Berk A, Lodish H, Darnell JE (2000). Biologie cellulaire moléculaire (4e éd.). New York : WH Freeman. ISBN 9780716737063.
- ^ un b “Traitement pré-ARNm eucaryote” . Académie Khan . Récupéré le 9 mars 2020 .
- ^ Jo BS, Choi SS (décembre 2015). “Introns : les avantages fonctionnels des introns dans les génomes” . Génomique & Informatique . 13 (4): 112–118. doi : 10.5808/GI.2015.13.4.112 . PMC 4742320 . PMID 26865841 .
- ^ “Étapes de traduction (article)” . Académie Khan . Récupéré le 10 mars 2020 .
- ^ “Noyau et ribosomes (article)” . Académie Khan . Récupéré le 10 mars 2020 .
- ^ un b Cooper GM (2000). La cellule : une approche moléculaire (2e éd.). Sunderland (Massachusetts) : Sinauer Associates. ISBN 9780878931064.
- ^ “Structure protéique : primaire, secondaire, tertiaire et quaternaire (article)” . Académie Khan . Récupéré le 11 mars 2020 .
- ^ Duan G, Walther D (février 2015). “Les rôles des modifications post-traductionnelles dans le contexte des réseaux d’interactions protéiques” . Biologie computationnelle PLOS . 11 (2) : e1004049. Bibcode : 2015PLSCB..11E4049D . doi : 10.1371/journal.pcbi.1004049 . PMC 4333291 . PMID 25692714 .
- ^ un b Schubert M, Walczak MJ, Aebi M, Wider G (juin 2015). “Modifications post-traductionnelles de protéines intactes détectées par spectroscopie RMN: application à la glycosylation”. Angewandte Chimie . 54 (24): 7096–7100. doi : 10.1002/anie.201502093 . PMID 25924827 .
- ^ Ciechanover A (janvier 2005). “Protéolyse: du lysosome à l’ubiquitine et au protéasome”. Revues naturelles. Biologie cellulaire moléculaire . 6 (1): 79–87. doi : 10.1038/nrm1552 . PMID 15688069 . S2CID 8953615 .
- ^ Brenner S, Miller JH (2001). Encyclopédie de la génétique . Elsevier Science Inc. p. 2800. ISBN 978-0-12-227080-2.
- ^ Murn J, Shi Y (août 2017). “Le chemin sinueux de la recherche sur la méthylation des protéines: jalons et nouvelles frontières”. Revues naturelles. Biologie cellulaire moléculaire . 18 (8): 517–527. doi : 10.1038/nrm.2017.35 . PMID 28512349 . S2CID 3917753 .
- ^ Drazic A, Myklebust LM, Ree R, Arnesen T (octobre 2016). “Le monde de l’acétylation des protéines” . Biochimica et Biophysica Acta (BBA) – Protéines et Protéomique . 1864 (10): 1372-1401. doi : 10.1016/j.bbapap.2016.06.007 . PMID 27296530 .
- ^ Bannister AJ, Kouzarides T (mars 2011). “Régulation de la chromatine par les modifications des histones” . Recherche cellulaire . 21 (3): 381–395. doi : 10.1038/cr.2011.22 . PMC 3193420 . PMID 21321607 .
- ^ un b Habara A, Steinberg MH (avril 2016). “Minireview : base génétique de l’hétérogénéité et de la gravité de la drépanocytose” . Biologie expérimentale et médecine . 241 (7): 689–696. doi : 10.1177/1535370216636726 . PMC 4950383 . PMID 26936084 .
- ^ Mangla, A.; Ehsan, M.; Agarwal, N.; En ligneMaruvada, S. (2020). “Anémie falciforme” . StatPearls . Édition StatPearls. PMID 29489205 . Récupéré le 12 mars 2020 .
- ^ Ilesanmi OO (janvier 2010). “Base pathologique des symptômes et des crises dans la drépanocytose : implications pour le conseil et la psychothérapie” . Rapports d’hématologie . 2 (1) : e2. doi : 10.4081/hr.2010.e2 . PMC 3222266 . PMID 22184515 .
- ^ un b “La Division Cellulaire, le Cancer | Apprenez la Science à Scitable” . www.nature.com . Récupéré le 30/11/2021 .
- ^ “p53, Cancer | Apprenez la science à Scitable” . www.nature.com . Récupéré le 30/11/2021 .
Liens externes
- Une vidéo utile visualisant le processus de conversion de l’ADN en protéine via la transcription et la traduction
- Vidéo visualisant le processus de repliement des protéines de la structure primaire non fonctionnelle à une structure protéique 3D mature et repliée en référence au rôle des mutations et du mauvais repliement des protéines dans la maladie
- Une vidéo plus poussée détaillant les différents types de modifications post-traductionnelles et leurs structures chimiques