ARN de transfert
L’ARN de transfert (en abrégé ARNt et anciennement appelé ARNs , pour ARN soluble [1] ) est une molécule adaptatrice composée d’ ARN , généralement de 76 à 90 Nucléotides de longueur (chez les eucaryotes), [2] qui sert de lien physique entre le ARNm et la séquence d’ acides aminés des protéines. L’ARN de transfert (ARNt) le fait en transportant un acide aminé vers la machinerie de synthèse des protéines d’une cellule appelée ribosome . Complémentation d’un Codon de 3 Nucléotides dans un ARN messager(ARNm) par un Anticodon à 3 Nucléotides de l’ARNt entraîne une synthèse protéique basée sur le code de l’ARNm. En tant que tels, les ARNt sont un composant nécessaire de la traduction , la synthèse biologique de nouvelles protéines conformément au code génétique .
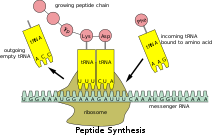 L’interaction de l’ARNt et de l’ARNm dans la synthèse des protéines.
L’interaction de l’ARNt et de l’ARNm dans la synthèse des protéines.
| ARNt | |
|---|---|
| Identifiants | |
| Symbole | t |
| Rfam | RF00005 |
| Autre informations | |
| Type d’ARN | gène , ARNt |
| Structures de l’ APB | PDBe 3icq, 1asy, 1asz, 1il2, 2tra, 3tra, 486d, 1fir, 1yfg, 3eph, 3epj, 3epk, 3epl, 1efw, 1c0a, 2ake, 2azx, 2dr2, 1f7u, 1f7v, 3foz, 2hgp, 2j00, 2ow82, , 2v46, 2v48, 2wdg, 2wdh, 2wdk, 2wdm, 2wh1 |
Aperçu
Alors que la séquence nucléotidique spécifique d’un ARNm spécifie quels acides aminés sont incorporés dans le produit protéique du gène à partir duquel l’ARNm est transcrit, le rôle de l’ARNt est de spécifier quelle séquence du code génétique correspond à quel acide aminé. [3] L’ARNm encode une protéine sous la forme d’une série de codons contigus, dont chacun est reconnu par un ARNt particulier. Une extrémité de l’ARNt correspond au code génétique dans une séquence de trois Nucléotides appelée Anticodon . L’Anticodon forme trois paires de bases complémentaires avec un Codon dans l’ARNm lors de la biosynthèse des protéines.
À l’autre extrémité de l’ARNt se trouve une liaison covalente à l’acide aminé qui correspond à la séquence de l’Anticodon. Chaque type de molécule d’ARNt ne peut être attaché qu’à un seul type d’acide aminé, de sorte que chaque organisme possède plusieurs types d’ARNt. Parce que le code génétique contient plusieurs codons qui spécifient le même acide aminé, il existe plusieurs molécules d’ARNt portant différents anticodons qui portent le même acide aminé.
La fixation covalente à l’ extrémité 3′ de l’ARNt est catalysée par des enzymes appelées aminoacyl ARNt synthétases . Au cours de la synthèse des protéines, les ARNt avec des acides aminés attachés sont délivrés au ribosome par des protéines appelées facteurs d’élongation , qui facilitent l’association de l’ARNt avec le ribosome, la synthèse du nouveau polypeptide et la translocation (mouvement) du ribosome le long de l’ARNm. Si l’Anticodon de l’ARNt correspond à l’ARNm, un autre ARNt déjà lié au ribosome transfère la chaîne polypeptidique en croissance de son extrémité 3 ‘à l’acide aminé attaché à l’extrémité 3’ de l’ARNt nouvellement délivré, une réaction catalysée par le ribosome. Un grand nombre de Nucléotides individuels dans une molécule d’ARNt peuvent êtrechimiquement modifié , souvent par méthylation ou désamidation . Ces bases inhabituelles affectent parfois l’interaction de l’ARNt avec les ribosomes et se produisent parfois dans l’ Anticodon pour modifier les propriétés d’appariement des bases. [4]
Structure
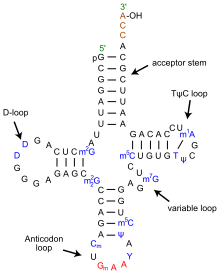
 Structure secondaire en trèfle de l’ARNt Phe de la levure.
Structure secondaire en trèfle de l’ARNt Phe de la levure. 
 Structure tertiaire de l’ARNt. Queue CCA en jaune, tige accepteur en violet, boucle variable en orange, bras D en rouge, bras Anticodon en bleu avec Anticodon en noir, bras T en vert.
Structure tertiaire de l’ARNt. Queue CCA en jaune, tige accepteur en violet, boucle variable en orange, bras D en rouge, bras Anticodon en bleu avec Anticodon en noir, bras T en vert. 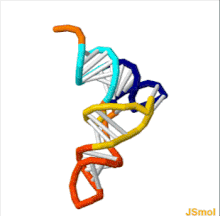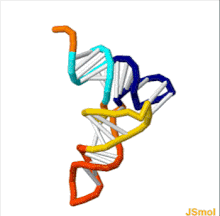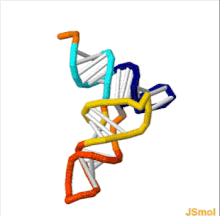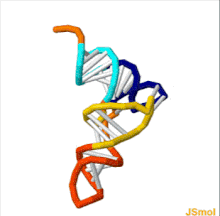
 GIF animé en 3D montrant la structure de la phénylalanine-ARNt de levure (PDB ID 1ehz). Les lignes blanches indiquent l’appariement des bases par des liaisons hydrogène. Dans l’orientation indiquée, la tige acceptrice est en haut et l’Anticodon en bas [5]
GIF animé en 3D montrant la structure de la phénylalanine-ARNt de levure (PDB ID 1ehz). Les lignes blanches indiquent l’appariement des bases par des liaisons hydrogène. Dans l’orientation indiquée, la tige acceptrice est en haut et l’Anticodon en bas [5]
La structure de l’ARNt peut être décomposée en sa Structure primaire , sa structure secondaire (généralement visualisée comme la structure en trèfle ) et sa Structure tertiaire [6] (tous les ARNt ont une structure 3D en forme de L similaire qui leur permet de s’intégrer dans le P et sites A du ribosome ). La structure en trèfle devient la structure en forme de L 3D par empilement coaxial des hélices, qui est un motif de Structure tertiaire d’ARN courant. Les longueurs de chaque bras, ainsi que le “diamètre” de la boucle, dans une molécule d’ARNt varient d’une espèce à l’autre. [6] [7]La structure de l’ARNt se compose des éléments suivants :
- Un groupe phosphate 5′-terminal .
- La tige acceptrice est une tige de 7 à 9 paires de bases (pb) constituée par l’appariement de bases du nucléotide 5′-terminal avec le nucléotide 3′-terminal (qui contient le groupe CCA 3′-terminal utilisé pour attacher l’amino acide). En général, de telles structures de type ARNt 3′-terminales sont appelées « étiquettes génomiques ». La tige acceptrice peut contenir des paires de bases non Watson-Crick. [6] [8]
- La queue CCA est une séquence cytosine – cytosine- adénine à l’extrémité 3 ‘de la molécule d’ARNt. L’acide aminé chargé sur l’ARNt par les aminoacyl ARNt synthétases , pour former l’ aminoacyl-ARNt , est lié de manière covalente au groupe 3′-hydroxyle sur la queue CCA. [9] Cette séquence est importante pour la reconnaissance de l’ARNt par les enzymes et critique dans la traduction. [10] [11] Chez les procaryotes, la séquence CCA est transcrite dans certaines séquences d’ARNt. Dans la plupart des ARNt procaryotes et des ARNt eucaryotes, la séquence CCA est ajoutée pendant le traitement et n’apparaît donc pas dans le gène de l’ARNt. [12]
- Le bras D est une tige de 4 à 6 pb se terminant par une boucle qui contient souvent de la dihydrouridine . [6]
- Le bras Anticodon est une tige de 5 pb dont la boucle contient l’ Anticodon . [6] La Structure primaire de l’ARNt 5′ à 3′ contient l’Anticodon mais dans l’ordre inverse, car la directionnalité 3′ à 5′ est nécessaire pour lire l’ARNm de 5′ à 3′.
- Le bras T est une tige de 4 à 5 pb se terminant par une boucle contenant la séquence TΨC où Ψ est la pseudouridine , une uridine modifiée . [6]
- Les bases qui ont été modifiées, notamment par méthylation (par exemple par l’ARNt (guanine-N7-)-méthyltransférase ), apparaissent en plusieurs positions dans l’ARNt. La première base d’Anticodon, ou position d’oscillation, est parfois modifiée en inosine (dérivé de l’adénine), queuosine (dérivé de la guanine), acide uridine-5-oxyacétique (dérivé de l’uracile), 5-méthylaminométhyl-2-thiouridine (dérivé de uracile), ou la lysidine (dérivée de la cytosine). [13]
Anticodon
Un Anticodon [14] est une unité de trois Nucléotides correspondant aux trois bases d’un Codon d’ ARNm . Chaque ARNt a une séquence distincte de triplet d’Anticodon qui peut former 3 paires de bases complémentaires à un ou plusieurs codons pour un acide aminé. Certains anticodons s’apparient avec plus d’un Codon en raison de l’ appariement des bases oscillantes . Fréquemment, le premier nucléotide de l’Anticodon est un nucléotide introuvable sur l’ARNm : l’ inosine , qui peut se lier par liaison hydrogène à plus d’une base dans la position de Codon correspondante. [4] : 29.3.9 Dans le code génétique , il est courant qu’un seul acide aminé soit spécifié par les quatre possibilités de troisième position, ou au moins par les pyrimidines et les purines ; par exemple, l’acide aminé glycine est codé par les séquences de codons GGU, GGC, GGA et GGG. D’autres Nucléotides modifiés peuvent également apparaître à la première position de l’Anticodon – parfois appelée «position oscillante» – entraînant des modifications subtiles du code génétique, comme par exemple dans les Mitochondries . [15]Par cellule, 61 types d’ARNt sont nécessaires pour fournir une correspondance biunivoque entre les molécules d’ARNt et les codons qui spécifient les acides aminés, car il existe 61 codons sens du code génétique standard. Cependant, de nombreuses cellules ont moins de 61 types d’ARNt car la base oscillante est capable de se lier à plusieurs, mais pas nécessairement à tous, des codons qui spécifient un acide aminé particulier. Au moins 31 ARNt sont nécessaires pour traduire, sans ambiguïté, les 61 codons sens. [3] [16]
Aminoacylation
L’ aminoacylation est le processus d’ajout d’un groupe aminoacyle à un composé. Il lie de manière covalente un acide aminé à l’extrémité CCA 3 ‘d’une molécule d’ARNt. Chaque ARNt est aminoacylé (ou chargé ) avec un acide aminé spécifique par une aminoacyl ARNt synthétase . Il existe normalement une seule aminoacyl ARNt synthétase pour chaque acide aminé, malgré le fait qu’il peut y avoir plus d’un ARNt et plus d’un Anticodon pour un acide aminé. La reconnaissance de l’ARNt approprié par les synthétases n’est pas médiée uniquement par l’Anticodon, et la tige acceptrice joue souvent un rôle de premier plan. [17] Réaction :
- acide aminé + ATP → aminoacyl-AMP + PPi
- aminoacyl-AMP + ARNt → aminoacyl-ARNt + AMP
Certains organismes peuvent avoir une ou plusieurs synthétases d’aminophosphate-ARNt manquantes. Cela conduit à la charge de l’ARNt par un acide aminé chimiquement apparenté, et grâce à l’utilisation d’une enzyme ou d’enzymes, l’ARNt est modifié pour être correctement chargé. Par exemple, Helicobacter pylori n’a pas de glutaminyl ARNt synthétase. Ainsi, la Glutamate ARNt synthétase charge l’ARNt-glutamine (ARNt-Gln) avec du Glutamate . Une amidotransférase convertit ensuite la chaîne latérale acide du Glutamate en amide, formant le gln-tRNA-Gln correctement chargé.
L’interférence avec l’aminoacylation peut être utile comme approche pour traiter certaines maladies : les cellules cancéreuses peuvent être relativement vulnérables à une aminoacylation perturbée par rapport aux cellules saines. La synthèse protéique associée au cancer et à la biologie virale est souvent très dépendante de molécules d’ARNt spécifiques. Par exemple, pour le cancer du foie, charger l’ARNt-Lys-CUU avec de la lysine soutient la croissance et les métastases des cellules cancéreuses du foie, tandis que les cellules saines dépendent beaucoup moins de cet ARNt pour soutenir la physiologie cellulaire. [18] De même, le virus de l’hépatite E nécessite un paysage d’ARNt qui diffère considérablement de celui associé aux cellules non infectées. [19]Par conséquent, l’inhibition de l’aminoacylation d’espèces d’ARNt spécifiques est considérée comme une nouvelle voie prometteuse pour le traitement rationnel d’une pléthore de maladies.
Liaison au ribosome
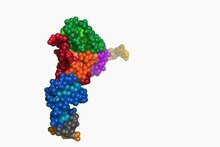
![]() Lire des médias La gamme de conformations adoptées par l’ARNt lorsqu’il transite par les sites A/T à travers les sites P/E sur le ribosome. Les codes de la Protein Data Bank (PDB) pour les modèles structurels utilisés comme points finaux de l’animation sont donnés. Les deux ARNt sont modélisés comme des ARNt spécifiques à la phénylalanine d’ Escherichia coli , avec l’ARNt A/T comme modèle d’homologie des coordonnées déposées. Codage couleur comme indiqué pour la Structure tertiaire de l’ARNt . Adapté de. [20]
Lire des médias La gamme de conformations adoptées par l’ARNt lorsqu’il transite par les sites A/T à travers les sites P/E sur le ribosome. Les codes de la Protein Data Bank (PDB) pour les modèles structurels utilisés comme points finaux de l’animation sont donnés. Les deux ARNt sont modélisés comme des ARNt spécifiques à la phénylalanine d’ Escherichia coli , avec l’ARNt A/T comme modèle d’homologie des coordonnées déposées. Codage couleur comme indiqué pour la Structure tertiaire de l’ARNt . Adapté de. [20]
Le ribosome a trois sites de liaison pour les molécules d’ARNt qui couvrent l’espace entre les deux sous-unités ribosomiques : les sites A (aminoacyle) , [21] P (peptidyle) et E (sortie) . De plus, le ribosome possède deux autres sites de liaison à l’ARNt qui sont utilisés lors du décodage de l’ARNm ou lors de l’initiation de la synthèse des protéines . Il s’agit du site T (appelé facteur d’élongation Tu ) et du site I (d’initiation). [22] [23] Par convention, les sites de liaison de l’ARNt sont désignés par le site sur la petite sous-unité ribosomique répertorié en premier et le site sur la grande sous-unité ribosomiqueclassé deuxième. Par exemple, le site A s’écrit souvent A/A, le site P, P/P, et le site E, E/E. [22] Les protéines de liaison comme L27, L2, L14, L15, L16 aux sites A et P ont été déterminées par marquage d’affinité par AP Czernilofsky et al. ( Proc. Natl. Acad. Sci, États-Unis , p. 230–234, 1974).
Une fois l’initiation de la traduction terminée, le premier ARNt aminoacyle est situé dans le site P/P, prêt pour le cycle d’élongation décrit ci-dessous. Au cours de l’élongation de la traduction, l’ARNt se lie d’abord au ribosome dans le cadre d’un complexe avec le facteur d’élongation Tu ( EF-Tu ) ou son homologue eucaryote ( eEF-1 ) ou archéen. Ce site initial de liaison à l’ARNt est appelé site A/T. Dans le site A/T, la moitié du site A réside dans la petite sous-unité ribosomale où se trouve le site de décodage de l’ARNm. Le site de décodage de l’ARNm est l’endroit où le Codon de l’ ARNm est lu pendant la traduction. La moitié du site T réside principalement sur la grande sous-unité ribosomiqueoù EF-Tu ou eEF-1 interagit avec le ribosome. Une fois le décodage de l’ARNm terminé, l’aminoacyl-ARNt est lié au site A/A et est prêt pour la prochaine liaison peptidique [24] à former avec son acide aminé attaché. Le peptidyl-ARNt, qui transfère le polypeptide en croissance à l’aminoacyl-ARNt lié au site A/A, est lié au site P/P. Une fois la liaison peptidique formée, l’ARNt dans le site P/P est acylé ou possède une extrémité 3′ libre , et l’ARNt dans le site A/A dissocie la chaîne polypeptidique en croissance. Pour permettre le cycle d’élongation suivant, les ARNt se déplacent ensuite à travers les sites de liaison hybrides A/P et P/E, avant de terminer le cycle et de résider dans les sites P/P et E/E. Une fois que les ARNt A/A et P/P se sont déplacés vers les sites P/P et E/E, l’ARNm s’est également déplacé d’uncodon et le site A/T est vacant, prêt pour le prochain cycle de décodage de l’ARNm. L’ARNt lié au site E/E quitte alors le ribosome.
Le site P/I est en fait le premier à se lier à l’ARNt aminoacyle, qui est délivré par un facteur d’initiation appelé IF2 chez les bactéries. [23] Cependant, l’existence du site P/I dans les ribosomes eucaryotes ou archéens n’a pas encore été confirmée. La protéine L27 du site P a été déterminée par marquage d’affinité par E. Collatz et AP Czernilofsky ( FEBS Lett. , Vol. 63, pp. 283-286, 1976).
Gènes d’ARNt
Les organismes varient dans le nombre de Gènes d’ARNt dans leur génome . Par exemple, le ver nématode C. elegans , un organisme modèle couramment utilisé dans les études génétiques , possède 29 647 [25] Gènes dans son génome nucléaire , dont 620 codent pour l’ARNt. [26] [27] La levure bourgeonnante Saccharomyces cerevisiae a 275 Gènes d’ARNt dans son génome.
Dans le génome humain, qui, selon les estimations de janvier 2013, compte environ 20 848 Gènes codant pour des protéines [28] au total, il existe 497 Gènes nucléaires codant pour des molécules d’ARNt cytoplasmiques et 324 Pseudogènes dérivés d’ARNt – des Gènes d’ARNt que l’on pense ne plus être fonctionnels [29] (bien qu’il ait été démontré que les pseudo-ARNt sont impliqués dans la résistance aux antibiotiques chez les bactéries). [30] Comme pour tous les eucaryotes, il existe 22 Gènes d’ARNt mitochondriaux [31] [ citation complète nécessaire ] chez l’homme. Des mutations dans certains de ces Gènes ont été associées à des maladies graves comme le syndrome MELAS . Régions dans le nucléairedes Chromosomes , très similaires en séquence aux Gènes d’ARNt mitochondriaux, ont également été identifiés (ressemblances d’ARNt). [32] Ces sosies d’ARNt sont également considérées comme faisant partie de l’ ADN mitochondrial nucléaire (Gènes transférés des Mitochondries au noyau). [32] [33] Le phénomène de multiples copies nucléaires d’ARNt mitochondrial (ARNt-sosie) a été observé dans de nombreux organismes supérieurs de l’homme à l’opossum [34] suggérant la possibilité que les sosies soient fonctionnels.
Les Gènes d’ARNt cytoplasmiques peuvent être regroupés en 49 familles selon leurs caractéristiques d’Anticodon. Ces Gènes se trouvent sur tous les Chromosomes, à l’exception des Chromosomes 22 et Y. Un clustering élevé sur 6p est observé (140 Gènes d’ARNt), ainsi que sur 1 chromosome. [29]
Le HGNC , en collaboration avec la base de données génomique des ARNt ( GtRNAdb ) et des experts dans le domaine, a approuvé des noms uniques pour les Gènes humains qui codent les ARNt.
Évolution
La moitié supérieure de l’ARNt (constituée du bras T et de la tige acceptrice avec le groupe phosphate 5′-terminal et le groupe CCA 3′-terminal) et la moitié inférieure (constituée du bras D et du bras Anticodon) sont des unités indépendantes en structure ainsi qu’en fonction. La moitié supérieure peut avoir évolué en premier, y compris l’étiquette génomique 3′-terminale qui, à l’origine, peut avoir marqué des molécules de type ARNt pour la réplication dans le monde de l’ARN précoce . La moitié inférieure peut avoir évolué plus tard en tant qu’expansion, par exemple lorsque la synthèse des protéines a commencé dans le monde de l’ARN et l’a transformé en un monde de ribonucléoprotéines (monde RNP ). Ce scénario proposé est appelé hypothèse d’étiquette génomique . En fait, les agrégats d’ARNt et de type ARNt ont une influence catalytique importante (c’est-à-dire que les ribozymes) sur la réplication encore aujourd’hui. Ces rôles peuvent être considérés comme des « fossiles moléculaires (ou chimiques) » du monde de l’ARN. [35]
Le contenu en ARNt génomique est une caractéristique de différenciation des génomes parmi les domaines biologiques de la vie : les archées présentent la situation la plus simple en termes de contenu en ARNt génomique avec un nombre uniforme de copies de Gènes, les bactéries ont une situation intermédiaire et les Eukarya présentent la situation la plus complexe. [36] Eukarya présente non seulement plus de contenu en Gènes d’ARNt que les deux autres royaumes, mais également une forte variation du nombre de copies de Gènes parmi différents isoaccepteurs, et cette complexité semble être due à des duplications de Gènes d’ARNt et à des changements dans la spécificité des anticodons [ citation nécessaire ] .
L’évolution du nombre de copies du gène de l’ARNt dans différentes espèces a été liée à l’apparition d’enzymes spécifiques de modification de l’ARNt (uridine méthyltransférases chez les bactéries et adénosine désaminases chez Eukarya), qui augmentent la capacité de décodage d’un ARNt donné. [36]Par exemple, l’ARNtAla code pour quatre isoaccepteurs d’ARNt différents (AGC, UGC, GGC et CGC). Chez Eukarya, les isoaccepteurs AGC sont extrêmement enrichis en nombre de copies de Gènes par rapport au reste des isoaccepteurs, et cela a été corrélé avec sa modification A-to-I de sa base oscillante. Cette même tendance a été montrée pour la plupart des acides aminés des espèces eucaryales. En effet, l’effet de ces deux modifications d’ARNt est également observé dans le biais d’utilisation des codons. Les Gènes fortement exprimés semblent être enrichis en codons qui utilisent exclusivement des codons qui seront décodés par ces ARNt modifiés, ce qui suggère un rôle possible de ces codons – et par conséquent de ces modifications d’ARNt – dans l’efficacité de la traduction. [36]
Il est important de noter que de nombreuses espèces ont perdu des ARNt spécifiques au cours de l’évolution. Par exemple, les mammifères et les oiseaux n’ont pas les mêmes 14 des 64 Gènes d’ARNt possibles, mais d’autres formes de vie contiennent ces ARNt. [37] Pour traduire les codons pour lesquels un ARNt d’appariement exact est manquant, les organismes ont recours à une stratégie appelée wobbling , dans laquelle des paires ARNt/ARNm imparfaitement appariées donnent toujours lieu à une traduction, bien que cette stratégie augmente également la propension aux erreurs de traduction. [38] Les raisons pour lesquelles les Gènes d’ARNt ont été perdus au cours de l’évolution restent à débattre, mais peuvent être liées à l’amélioration de la résistance à l’infection virale. [39]Étant donné que les triplets de Nucléotides peuvent présenter plus de combinaisons qu’il n’y a d’acides aminés et d’ARNt associés, il existe une redondance dans le code génétique et plusieurs codons différents de 3 Nucléotides peuvent exprimer le même acide aminé. Ce biais de codons est ce qui nécessite une optimisation des codons, et en examinant les séquences codantes du répertoire d’ARNt de votre organisme cible, vous pouvez échanger des codons en fonction de votre système d’expression. [40]
fragments dérivés d’ARNt
Les fragments dérivés d’ARNt (ou tRF) sont de courtes molécules qui émergent après clivage des ARNt matures ou du transcrit précurseur. [41] [42] [43] [44] Les ARNt cytoplasmiques et mitochondriaux peuvent produire des fragments. [45] Il existe au moins quatre types structuraux de tRF que l’on pense provenir d’ARNt matures, y compris les moitiés d’ARNt relativement longues et les 5′-tRF, 3′-tRF et i-tRF courts. [41] [45] [46] L’ARNt précurseur peut être clivé pour produire des molécules à partir des séquences 5′ leader ou 3′ trail. Les enzymes de clivage comprennent l’angiogénine, le Dicer, la RNase Z et la RNase P. [41] [42]En particulier dans le cas de l’angiogénine, les tRF ont un phosphate cyclique caractéristique inhabituel à leur extrémité 3′ et un groupe hydroxyle à l’extrémité 5′. [47] Les tRF semblent jouer un rôle dans l’interférence ARN , en particulier dans la suppression des rétrovirus et des rétrotransposons qui utilisent l’ARNt comme amorce pour la réplication. Les demi-ARNt clivés par l’angiogénine sont également appelés ARNti. La biogenèse de fragments plus petits, y compris ceux qui fonctionnent comme des PiARN , est moins bien comprise. [48]
les tRF ont de multiples dépendances et rôles ; comme présenter des changements significatifs entre les sexes, entre les races et l’état de la maladie. [45] [49] [50] Fonctionnellement, ils peuvent être chargés sur Ago et agir par les voies ARNi, [43] [46] [51] participer à la formation de granules de stress, [52] déplacer les ARNm des protéines de liaison à l’ARN [53] ou inhiber la traduction. [54] Au niveau du système ou de l’organisme, les quatre types de tRF ont un large éventail d’activités. Fonctionnellement, les tRF sont associés à une infection virale, [55] au cancer, [46] à la prolifération cellulaire [47]et aussi avec la régulation épigénétique transgénérationnelle du métabolisme. [56]
Les tRF ne se limitent pas aux humains et il a été démontré qu’ils existent dans plusieurs organismes. [46] [57] [58] [59]
Deux outils en ligne sont disponibles pour ceux qui souhaitent en savoir plus sur les tRF : le framework for the interactive exploration of mi tochondrial and n uclear t RNA fragments ( MINTbase ) [60] [61] et la base de données relationnelle de T ransfer R NA related F ragments ( tRFdb ). [62] MINTbase fournit également un schéma de dénomination pour la dénomination des tRF appelés plaques d’immatriculation tRF (ou MINTcodes) qui est indépendant du génome ; le schéma comprime une séquence d’ARN en une chaîne plus courte.
ARNt modifiés
Les ARNt allongeurs suppresseurs artificiels sont utilisés pour incorporer des acides aminés non naturels à des codons non-sens placés dans la séquence codante d’un gène. Des ARNt initiateurs modifiés (ARNt fMet2 avec Anticodon CUA codé par le gène metY ) ont été utilisés pour initier la traduction au niveau du Codon d’arrêt ambre UAG. Ce type d’ARNt modifié est appelé ARNt suppresseur de non-sens car il supprime le signal d’arrêt de la traduction qui se produit normalement au niveau des codons UAG. L’ARNt initiateur ambre insère la méthionine [63] et la glutamine [64] au niveau des codons UAG précédés d’une forte Séquence Shine-Dalgarno. Une enquête sur l’ARNt initiateur ambre a montré qu’il était orthogonal au Codon de départ AUG régulier ne montrant aucun événement d’initiation de traduction hors cible détectable dans une souche d’ E. coli recodée génomiquement . [63]
biogenèse de l’ARNt
Dans les cellules eucaryotes , les ARNt sont transcrits par l’ARN polymérase III sous forme de pré-ARNt dans le noyau. [65] L’ARN polymérase III reconnaît deux séquences promotrices en aval hautement conservées : la région de contrôle intragénique 5′ (5′-ICR, région de contrôle D ou boîte A) et la région 3′-ICR (région de contrôle T ou boîte B). ) à l’intérieur des Gènes d’ARNt. [2] [66] [67] Le premier promoteur commence à +8 des ARNt matures et le deuxième promoteur est situé à 30–60 Nucléotides en aval du premier promoteur. La transcription se termine après une séquence de quatre thymidines ou plus . [2] [67]
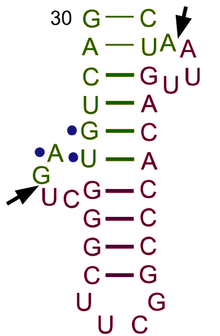
 Motif renflement-hélice-renflement d’un intron d’ARNt
Motif renflement-hélice-renflement d’un intron d’ARNt
Les pré-ARNt subissent des modifications importantes à l’intérieur du noyau. Certains pré-ARNt contiennent des introns qui sont épissés ou coupés pour former la molécule d’ARNt fonctionnelle; [68] chez les bactéries, ces auto- épissages , alors que chez les eucaryotes et les archées , ils sont éliminés par des endonucléases d’épissage d’ARNt . [69] Le pré-ARNt eucaryote contient un motif de structure renflement-hélice-renflement (BHB) qui est important pour la reconnaissance et l’épissage précis de l’intron d’ARNt par les endonucléases. [70] Cette position et cette structure de motif sont conservées de manière évolutive. Cependant, certains organismes, tels que les algues unicellulaires, ont une position non canonique du motif BHB ainsi que les extrémités 5 ‘et 3’ de la séquence d’intron épissée.[70] La séquence 5′ est éliminée par la RNase P , [71] tandis que l’extrémité 3′ est éliminée par l’enzyme tRNase Z. [72] Une exception notable est dans l’ archaeon Nanoarchaeum equitans , qui ne possède pas d’enzyme RNase P et a un promoteur placé de telle sorte que la transcription commence à l’extrémité 5 ‘de l’ARNt mature. [73] La queue 3′ CCA sans matrice est ajoutée par une nucléotidyl transférase . [74] Avant que les ARNt ne soient exportés dans le cytoplasme par Los1/ Xpo-t , [75] [76] les ARNt sont aminoacylés. [77] L’ordre des événements de traitement n’est pas conservé. Par exemple, chez la levure , l’épissage ne s’effectue pas dans le noyau mais du côté cytoplasmique des membranes mitochondriales . [78]
Néanmoins, en mars 2021, des chercheurs ont rapporté des preuves suggérant qu’une forme préliminaire d’ARN de transfert aurait pu être une molécule réplicatrice au tout début du développement de la vie, ou abiogenèse . [79] [80]
Histoire
L’existence de l’ARNt a été émise pour la première fois par Francis Crick , sur la base de l’hypothèse qu’il doit exister une molécule adaptatrice capable de médier la traduction de l’alphabet ARN en alphabet protéique. Paul C Zamecnik et Mahlon Hoagland ont découvert l’ARNt [81] Des recherches importantes sur la structure ont été menées au début des années 1960 par Alex Rich et Don Caspar , deux chercheurs à Boston, le groupe Jacques Fresco de l’Université de Princeton et un groupe du Royaume-Uni au King’s College de Londres . [82] En 1965, Robert W. Holley de l’Université Cornellrapporté la Structure primaire et suggéré trois structures secondaires. [83] L’ARNt a été cristallisé pour la première fois à Madison, Wisconsin, par Robert M. Bock. [84] La structure en trèfle a été confirmée par plusieurs autres études dans les années suivantes [85] et a finalement été confirmée à l’aide d’études de cristallographie aux rayons X en 1974. Deux groupes indépendants, Kim Sung-Hou travaillant sous Alexander Rich et un groupe britannique dirigé par Aaron Klug , a publié les mêmes résultats de cristallographie en un an. [86] [87]
Voir également
- Modèle de trèfle d’ARNt
- Kim Sung Hou
- Embrasser tige-boucle
- ARNm
- ARN non codant et introns
- Séquence glissante
- ARNtm
- Transfert de structures de type ARN
- Traduction
- tRNADB
- Hypothèse d’oscillation
- Aminoacyl-ARNt
Références
- ^ Plescia OJ, Palczuk NC, Cora-Figueroa E, Mukherjee A, Braun W (octobre 1965). “Production d’anticorps contre l’ARN soluble (ARNs)” . Actes de l’Académie nationale des sciences des États-Unis d’Amérique . 54 (4): 1281-1285. Bibcode : 1965PNAS…54.1281P . doi : 10.1073/pnas.54.4.1281 . PMC 219862 . PMID 5219832 .
- ^ un bc Sharp SJ, Schaack J, Cooley L, Burke DJ, Söll D (1985). “Structure et transcription des Gènes d’ARNt eucaryotes”. Examens critiques du CRC en biochimie . 19 (2): 107-144. doi : 10.3109/10409238509082541 . PMID 3905254 .
- ^ un b Crick FH (décembre 1968). “L’origine du code génétique”. Tourillon de biologie moléculaire . 38 (3): 367–379. doi : 10.1016/0022-2836(68)90392-6 . PMID 4887876 .
- ^ un b Stryer L, Berg JM, Tymoczko JL (2002). Biochimie (5e éd.). San Francisco : WH Freeman. ISBN 978-0-7167-4955-4.
- ^ ARN %28tARN%29 “ARN de transfert (ARNt)” . Proteopedia.org . Récupéré le 7 novembre 2018 . {{cite web}}: Vérifier |url=la valeur ( aide )
- ^ un bcdef Itoh Y , Sekine S, Suetsugu S, Yokoyama S (juillet 2013). “Structure tertiaire de l’ARNt de sérénocystéine bactérienne” . Recherche sur les acides nucléiques . 41 (13): 6729–6738. doi : 10.1093/nar/gkt321 . PMC 3711452 . PMID 23649835 .
- ^ Goodenbour JM, Pan T (29 octobre 2006). “Diversité des Gènes d’ARNt chez les eucaryotes” . Recherche sur les acides nucléiques . 34 (21): 6137–6146. doi : 10.1093/nar/gkl725 . PMC 1693877 . PMID 17088292 .
- ^ Jahn M, Rogers MJ, Söll D (juillet 1991). “Les Nucléotides de tige d’Anticodon et d’accepteur dans l’ARNt (Gln) sont des éléments de reconnaissance majeurs pour la glutaminyl-ARNt synthétase d’E. coli”. Nature . 352 (6332): 258–260. Bibcode : 1991Natur.352..258J . doi : 10.1038/352258a0 . PMID 1857423 . S2CID 4263705 .
- ^ Ibba M, Soll D (juin 2000). “Synthèse d’aminoacyl-ARNt”. Revue annuelle de biochimie . 69 (1): 617–650. doi : 10.1146/annurev.biochem.69.1.617 . PMID 10966471 .
- ^ Sprinzl M, Cramer F (1979). “L’extrémité -CCA de l’ARNt et son rôle dans la biosynthèse des protéines”. Progrès dans la recherche sur les acides nucléiques et la biologie moléculaire . 22 : 1–69. doi : 10.1016/s0079-6603(08)60798-9 . ISBN 978-0-12-540022-0. PMID 392600 .
- ^ Vert R, Noller HF (1997). “Ribosomes et traduction”. Revue annuelle de biochimie . 66 : 679–716. doi : 10.1146/annurev.biochem.66.1.679 . PMID 9242921 .
- ^ Aebi M, Kirchner G, Chen JY, Vijayraghavan U, Jacobson A, Martin NC, Abelson J, et al. (septembre 1990). “Isolement d’un mutant sensible à la température avec une nucléotidyltransférase d’ARNt modifiée et clonage du gène codant pour la nucléotidyltransférase d’ARNt dans la levure Saccharomyces cerevisiae” . Le Journal de chimie biologique . 265 (27): 16216–16220. doi : 10.1016/S0021-9258(17)46210-7 . PMID 2204621 .
- ^ McCloskey JA, Nishimura S (novembre 1977). “Nucléosides modifiés dans l’ARN de transfert”. Comptes de la recherche chimique . 10 (11): 403–410. doi : 10.1021/ar50119a004 .
- ^ Felsenfeld G, Cantoni GL (mai 1964). “Utilisation d’études de dénaturation thermique pour étudier la séquence de bases de l’ARNs de sérine de levure” . Actes de l’Académie nationale des sciences des États-Unis d’Amérique . 51 (5): 818–826. Bibcode : 1964PNAS…51..818F . doi : 10.1073/pnas.51.5.818 . PMC 300168 . PMID 14172997 .
- ^ Suzuki T, Suzuki T (juin 2014). “Un paysage complet des modifications post-transcriptionnelles dans les ARNt mitochondriaux de mammifères” . Recherche sur les acides nucléiques . 42 (11): 7346–7357. doi : 10.1093/nar/gku390 . PMC 4066797 . PMID 24831542 .
- ^ Lodish H, Berk A, Matsudaira P, Kaiser CA, Krieger M, Scott MP, Zipursky SL, Darnell J. (2004). Biologie Moléculaire de la Cellule . WH Freeman : New York. 5e éd. [ ISBN manquant ] [ page nécessaire ]
- ^ Schimmel P, Giegé R, Moras D, Yokoyama S (octobre 1993). “Un code ARN opérationnel pour les acides aminés et une relation possible avec le code génétique” . Actes de l’Académie nationale des sciences des États-Unis d’Amérique . 90 (19): 8763–8768. Bibcode : 1993PNAS…90.8763S . doi : 10.1073/pnas.90.19.8763 . PMC 47440 . PMID 7692438 .
- ^ Zhang R, Noordam L, Ou X, Ma B, Li Y, Das P, Shi S, Liu J, Wang L, Li P, Verstegen MM, Reddy DS, van der Laan LJ, Peppelenbosch MP, Kwekkeboom J, Smits R , Pan Q (janvier 2021). “Le processus biologique de charge de la lysine-ARNt est thérapeutiquement ciblable dans le cancer du foie” . Foie Int . 41 (1): 206–219. doi : 10.1111/liv.14692 . PMC 7820958 . PMID 33084231 .
- ^ Ou X, Ma B, Zhang R, Miao Z, Cheng A, Peppelenbosch MP, Pan Q (juin 2020). “Une méthode qPCR simplifiée révélant le remodelage de l’ARNt lors d’une infection par le virus de l’hépatite E de génotype 3” . Lettres FEBS . 594 (12): 2005–2015. doi : 10.1002/1873-3468.13764 . PMID 32133647 .
- ^ Dunkle JA, Wang L, Feldman MB, Pulk A, Chen VB, Kapral GJ, Noeske J, Richardson JS, Blanchard SC, Cate JH (mai 2011). “Structures du ribosome bactérien dans les états classiques et hybrides de la liaison de l’ARNt” . Sciences . 332 (6032): 981–984. Bibcode : 2011Sci…332..981D . doi : 10.1126/science.1202692 . PMC 3176341 . PMID 21596992 .
- ^ Konevega AL, Soboleva NG, Makhno VI, Semenkov YP, Wintermeyer W, Rodnina MV, Katunin VI (janvier 2004). “Les bases puriques en position 37 de l’ARNt stabilisent l’interaction Codon–Anticodon dans le site ribosomal A par empilement et interactions dépendantes de Mg2+” . ARN . 10 (1): 90–101. doi : 10.1261/rna.5142404 . PMC 1370521 . PMID 14681588 .
- ^ un b Agirrezabala X, Frank J (août 2009). “Elongation en traduction en tant qu’interaction dynamique entre le ribosome, l’ARNt et les facteurs d’élongation EF-G et EF-Tu” . Revues trimestrielles de biophysique . 42 (3): 159–200. doi : 10.1017/S0033583509990060 . PMC 2832932 . PMID 20025795 .
- ^ un b Allen GS, Zavialov A, Gursky R, Ehrenberg M, Frank J (juin 2005). “La structure cryo-EM d’un complexe d’initiation de la traduction d’Escherichia coli” . Cellule . 121 (5): 703–712. doi : 10.1016/j.cell.2005.03.023 . PMID 15935757 . S2CID 16146867 .
- ^ Tirumalai MR, Rivas M, Tran Q, Fox GE (novembre 2021). “Le Centre de Peptidyl Transferase : une Fenêtre au Passé”. Microbiol Mol Biol Rév . 85 (4) : e0010421. doi : 10.1128/MMBR.00104-21 . PMC 8579967. PMID 34756086 .
- ^ Site Web de WormBase, http://www.wormbase.org Archivé le 20/04/2017 sur la Wayback Machine , version WS187, date du 25 janvier 2008.
- ^ Spieth J, Lawson D (janvier 2006). “Vue d’ensemble de la structure des Gènes” . WormBook : 1–10. doi : 10.1895/wormbook.1.65.1 . PMC 4781370 . PMID 18023127 .
- ^ Hartwell LH, Hood L, Goldberg ML, Reynolds AE, Silver LM, Veres RC. (2004). Génétique : des Gènes aux génomes 2e éd. McGraw Hill : New York. p. 264.
- ^ Ensembl version 70 – janvier 2013 http://www.ensembl.org/Homo_sapiens/Info/StatsTable?db=core Archivé le 15/12/2013 sur la Wayback Machine
- ^ un b Lander ES, Linton LM, Birren B, Nusbaum C, Zody MC, Baldwin J, et al. (Consortium international de séquençage du génome humain) (février 2001). “Séquençage initial et analyse du génome humain” (PDF) . Nature . 409 (6822): 860–921. Bibcode : 2001Natur.409..860L . doi : 10.1038/35057062 . PMID 11237011 .
- ^ Rogers TE, Ataide SF, Dare K, Katz A, Seveau S, Roy H, Ibba M (2012). “Un pseudo-ARNt module la résistance aux antibiotiques chez Bacillus cereus” . PLOS ONE . 7 (7) : e41248. Bibcode : 2012PLoSO…741248R . doi : 10.1371/journal.pone.0041248 . PMC 3399842 . PMID 22815980 .
- ^ Idem. p. 529.
- ^ un b Telonis AG, Loher P, Kirino Y, Rigoutsos I (2014). “Sosies d’ARNt nucléaires et mitochondriaux dans le génome humain” . Frontières en génétique . 5 : 344. doi : 10.3389/fgene.2014.00344 . PMC 4189335 . PMID 25339973 .
- ^ Ramos A, Barbena E, Mateiu L, del Mar González M, Mairal Q, Lima M, Montiel R, Aluja MP, Santos C, et al. (novembre 2011). « Insertions nucléaires d’origine mitochondriale : mise à jour de la base de données et utilité dans les études sur le cancer ». Mitochondrie . 11 (6): 946–953. doi : 10.1016/j.mito.2011.08.009 . PMID 21907832 .
- ^ Telonis AG, Kirino Y, Rigoutsos I (2015). “Sosies d’ARNt mitochondriaux dans les Chromosomes nucléaires : pourraient-ils être fonctionnels ?” . ARN Biol . 12 (4): 375–380. doi : 10.1080/15476286.2015.1017239 . PMC 4615777 . PMID 25849196 .
- ^ Nancy Maizels et Alan M. Weiner: L’hypothèse de l’étiquette génomique – Ce que les fossiles moléculaires nous disent sur l’évolution de l’ARNt , dans: Le monde de l’ARN, deuxième édition. 1999 Cold Spring Harbor Laboratory Press ISBN 978-0-87969-561-3 / 99, PDF
- ^ un bc Novoa EM, Pavon-Eternod M, Pan T, Ribas de Pouplana L (mars 2012). “Un rôle pour les modifications d’ARNt dans la structure du génome et l’utilisation des codons” . Cellule . 149 (1): 202–213. doi : 10.1016/j.cell.2012.01.050 . PMID 22464330 . S2CID 16487609 .
- ^ Ou X, Peng W, Yang Z, Cao J, Wang M, Peppelenbosch MP, Pan Q, Cheng A (novembre 2020). “Gènes d’ARNt manquants et conservés de manière évolutive chez l’homme et l’aviaire” . Infecter. Genet. Évol . 85 : 104460. doi : 10.1016/j.meegid.2020.104460 . PMID 32679345 .
- ^ Ou X, Cao J, Cheng A, Peppelenbosch MP, Pan Q (mars 2019). “Erreurs dans le décodage traductionnel : oscillation de l’ARNt ou mauvaise incorporation ?” . Génétique PLOS . 15 (3): 2979-2986. doi : 10.1371/journal.pgen.1008017 . PMC 3158919 . PMID 21930591 .
- ^ Ou X, Wang M, Mao S, Cao J, Cheng A, Zhu D, Chen S, Jia R, Liu M, Yang Q, Wu Y, Zhao X, Zhang S, Liu Y, Yu Y, Zhang L, Chen X, député de Peppelenbosch, Pan Q (juillet 2018). “La traduction incompatible entraîne une évolution convergente et une atténuation virale lors du développement d’un vaccin vivant atténué” . Devant. Cellule. Infecter. Microbiol . 8 : 249. doi : 10.3389/fcimb.2018.00249 . PMC 6058041 . PMID 30073153 .
- ^ van Warmerdam, T. “YourBioHelper.com” . VotreBioHelper . {{cite web}}: Maint CS1 : url-status ( lien )
- ^ un bc Gebetsberger J, Polacek N (décembre 2013). « Couper en tranches les ARNt pour stimuler la diversité fonctionnelle des ARNnc » . Biologie de l’ARN . 10 (12): 1798–1806. doi : 10.4161/rna.27177 . PMC 3917982 . PMID 24351723 .
- ^ un b Shigematsu M, Honda S, Kirino Y (2014). “ARN de transfert comme source de petit ARN fonctionnel” . Journal de biologie moléculaire et d’imagerie moléculaire . 1 (2): 8. PMC 4572697 . PMID 26389128 .
- ^ un b Sobala A, Hutvagner G (2011). “Fragments dérivés d’ARN de transfert: origines, traitement et fonctions” (PDF) . Examens interdisciplinaires de Wiley : ARN . 2 (6): 853–862. doi : 10.1002/wrna.96 . manche : 10453/18187 . PMID 21976287 . S2CID 206554146 .
- ^ Keam SP, Hutvagner G (novembre 2015). “Fragments dérivés d’ARNt (tRF) : nouveaux rôles émergents pour un ARN ancien dans la régulation de l’expression génique” . La vie . 5 (4): 1638–1651. doi : 10.3390/life5041638 . PMC 4695841 . PMID 26703738 .
- ^ un bc Telonis AG, Loher P, Honda S, Jing Y, Palazzo J, Kirino Y, Rigoutsos I (juillet 2015). “La dissection des complexités des fragments dérivés d’ARNt à l’aide de transcriptomes personnalisés révèle de nouvelles classes de fragments et des dépendances inattendues” . Oncocible . 6 (28): 24797–822. doi : 10.18632/oncotarget.4695 . PMC 4694795 . PMID 26325506 .
- ^ un bcd Kumar P, Anaya J, Mudunuri SB, Dutta A (octobre 2014). “La méta-analyse de fragments d’ARN dérivés d’ARNt révèle qu’ils sont conservés au cours de l’évolution et s’associent aux protéines AGO pour reconnaître des cibles d’ARN spécifiques” . BMC Biologie . 12 : 78. doi : 10.1186/s12915-014-0078-0 . PMC 4203973 . PMID 25270025 .
- ^ a b Honda S, Loher P, Shigematsu M, Palazzo JP, Suzuki R, Imoto I, Rigoutsos I, Kirino Y (juillet 2015). “Les moitiés d’ARNt dépendantes des hormones sexuelles améliorent la prolifération cellulaire dans les cancers du sein et de la prostate” . Actes de l’Académie nationale des sciences des États-Unis d’Amérique . 112 (29) : E3816–E3825. Bibcode : 2015PNAS..112E3816H . doi : 10.1073/pnas.1510077112 . PMC 4517238 . PMID 26124144 .
- ^ Schorn, AJ; Martienssen, R (octobre 2018). “Tie-Break : l’hôte et les rétrotransposons jouent l’ARNt” . Tendances en biologie cellulaire . 28 (10): 793–806. doi : 10.1016/j.tcb.2018.05.006 . PMC 6520983 . PMID 29934075 .
- ^ Telonis AG, Rigoutsos I (mars 2018). “Disparités raciales dans la contribution des isoformes de miARN et des fragments dérivés d’ARNt au cancer du sein triple négatif” . Cancer Res . 78 (5) : 1140–1154. doi : 10.1158/0008-5472.CAN-17-1947 . PMC 5935570 . PMID 29229607 .
- ^ Telonis AG, Loher P, Magee R, Pliatsika V, Londin E, Kirino Y, Rigoutsos I (juin 2019). “Les fragments d’ARNt montrent un entrelacement avec des ARNm de contenu de répétition spécifique et ont des liens avec des disparités” . Cancer Res . 79 (12): 3034–49. doi : 10.1158/0008-5472.CAN-19-0789 . PMC 6571059 . PMID 30996049 .
- ^ Shigematsu M, Kirino Y (2015). “ARNt court non codant dérivé d’ARNt en tant que partenaires d’interaction des protéines Argonaute” . Régulation des Gènes et biologie des systèmes . 9 : 27–33. doi : 10.4137/GRSB.S29411 . PMC 4567038 . PMID 26401098 .
- ^ Emara MM, Ivanov P, Hickman T, Dawra N, Tisdale S, Kedersha N, Hu GF, Anderson P (avril 2010). “Les ARN induits par le stress dérivés de l’ARNt induits par l’angiogénine favorisent l’assemblage des granules de stress induit par le stress” . Le Journal de chimie biologique . 285 (14): 10959–10968. doi : 10.1074/jbc.M109.077560 . PMC 2856301 . PMID 20129916 .
- ^ Goodarzi H, Liu X, Nguyen HC, Zhang S, Fish L, Tavazoie SF (mai 2015). “Les fragments endogènes dérivés d’ARNt suppriment la progression du cancer du sein via le déplacement YBX1” . Cellule . 161 (4): 790–802. doi : 10.1016/j.cell.2015.02.053 . PMC 4457382 . PMID 25957686 .
- ^ Ivanov P, Emara MM, Villen J, Gygi SP, Anderson P (août 2011). “Les fragments d’ARNt induits par l’angiogénine inhibent l’initiation de la traduction” . Cellule moléculaire . 43 (4): 613–623. doi : 10.1016/j.molcel.2011.06.022 . PMC 3160621 . PMID 21855800 .
- ^ Selitsky SR, Baran-Gale J, Honda M, Yamane D, Masaki T, Fannin EE, Guerra B, Shirasaki T, Shimakami T, Kaneko S, Lanford RE, Lemon SM, Sethupathy P (janvier 2015). “Les petits ARN dérivés d’ARNt sont plus nombreux et plus abondants que les microARN dans les hépatites chroniques B et C” . Rapports scientifiques . 5 : 7675. Bibcode : 2015NatSR…5E7675S . doi : 10.1038/srep07675 . PMC 4286764 . PMID 25567797 .
- ^ Sharma U, Conine CC, Shea JM, Boskovic A, Derr AG, Bing XY, Belleannee C, Kucukural A, Serra RW, Sun F, Song L, Carone BR, Ricci EP, Li XZ, Fauquier L, Moore MJ, Sullivan R, Mello CC, Garber M, Rando OJ (janvier 2016). “Biogenèse et fonction des fragments d’ARNt pendant la maturation et la fécondation des spermatozoïdes chez les mammifères” . Sciences . 351 (6271): 391–396. Bib code : 2016Sci …351..391S . doi : 10.1126/science.aad6780 . PMC 4888079 . PMID 26721685 .
- ^ Casas E, Cai G, Neill JD (2015). “Caractérisation des fragments d’ARN dérivés de l’ARN de transfert circulant chez les bovins” . Frontières en génétique . 6 : 271. doi : 10.3389/fgene.2015.00271 . PMC 4547532 . PMID 26379699 .
- ^ Hirose Y, Ikeda KT, Noro E, Hiraoka K, Tomita M, Kanai A (juillet 2015). “Cartographie précise et dynamique des fragments dérivés d’ARNt (tRF) dans le développement de Triops cancriformis (crevette têtard)” . Génétique BMC . 16 : 83. doi : 10.1186/s12863-015-0245-5 . PMC 4501094 . PMID 26168920 .
- ^ Karaiskos S, Naqvi AS, Swanson KE, Grigoriev A (septembre 2015). “Modulation induite par l’âge des fragments dérivés d’ARNt chez la drosophile et leurs cibles potentielles” . Biologie directe . 10 : 51. doi : 10.1186/s13062-015-0081-6 . PMC 4572633 . PMID 26374501 .
- ^ Pliatsika V, Loher P, Telonis AG, Rigoutsos I (août 2016). “MINTbase : un cadre pour l’exploration interactive des fragments d’ARNt mitochondriaux et nucléaires” . Bioinformatique . 32 (16): 2481–2489. doi : 10.1093/bioinformatique/btw194 . PMC 4978933 . PMID 27153631 .
- ^ Pliatsika V, Loher P, Magee R, Telonis AG, Londin E, Shigematsu M, Kirino Y, Rigoutsos I (janvier 2018). “MINTbase v2.0 : une base de données complète pour les fragments dérivés d’ARNt qui comprend des fragments nucléaires et mitochondriaux de tous les projets de l’Atlas du génome du cancer” . Recherche sur les acides nucléiques . 46(D1) (D1) : D152–D159. doi : 10.1093/nar/gkx1075 . PMC 5753276 . PMID 29186503 .
- ^ Kumar P, Mudunuri SB, Anaya J, Dutta A (janvier 2015). “tRFdb : une base de données pour les fragments d’ARN de transfert” . Recherche sur les acides nucléiques . 43 (Numéro de base de données) : D141-5. doi : 10.1093/nar/gku1138 . PMC 4383946 . PMID 25392422 .
- ^ un b Vincent RM, Wright BW, Jaschke PR (avril 2019). “Mesurer l’orthogonalité de l’ARNt initiateur d’ambre dans un organisme génomiquement enregistré”. ACS Biologie Synthétique . 8 (4): 675–685. doi : 10.1021/acssynbio.9b00021 . PMID 30856316 . S2CID 75136654 .
- ^ Govindan A, Miryala S, Mondal S, Varshney U (novembre 2018). “Développement de systèmes d’analyse pour le décodage des codons ambrés aux étapes d’initiation et d’allongement chez les mycobactéries” . Journal de bactériologie . 200 (22). doi : 10.1128/jb.00372-18 . PMC 6199473 . PMID 30181124 .
- ^ RJ blanc (mars 1997). “Régulation des ARN polymérases I et III par la protéine du rétinoblastome : un mécanisme de contrôle de la croissance ?”. Tendances en sciences biochimiques . 22 (3): 77–80. doi : 10.1016/S0968-0004(96)10067-0 . PMID 9066256 .
- ^ Sharp S, Dingermann T, Söll D (septembre 1982). “Les séquences intragéniques minimales requises pour la promotion de la transcription du gène de l’ARNt eucaryote” . Recherche sur les acides nucléiques . 10 (18): 5393–5406. doi : 10.1093/nar/10.18.5393 . PMC 320884 . PMID 6924209 .
- ^ un b Dieci G, Fiorino G, Castelnuovo M, Teichmann M, Pagano A (décembre 2007). “Le transcriptome de l’ARN polymérase III en expansion”. Tendances en génétique . 23 (12): 614–622. doi : 10.1016/j.tig.2007.09.001 . hdl : 11381/1706964 . PMID 17977614 .
- ^ Tocchini-Valentini GD, Fruscoloni P, Tocchini-Valentini GP (décembre 2009). “Traitement de pretRNA contenant plusieurs introns” . Actes de l’Académie nationale des sciences des États-Unis d’Amérique . 106 (48): 20246–20251. Bibcode : 2009PNAS..10620246T . doi : 10.1073/pnas.0911658106 . PMC 2787110 . PMID 19910528 .
- ^ Abelson J, Trotta CR, Li H (mai 1998). “épissage d’ARNt” . Le Journal de chimie biologique . 273 (21): 12685–12688. doi : 10.1074/jbc.273.21.12685 . PMID 9582290 .
- ^ un b Soma A (2014). “Gènes d’ARNt circulairement permutés: leur expression et leurs implications pour leur pertinence et leur développement physiologiques” . Frontières en génétique . 5 : 63. doi : 10.3389/fgene.2014.00063 . PMC 3978253 . PMID 24744771 .
- ^ Frank DN, Pace NR (1998). “Ribonucléase P : unité et diversité dans un ribozyme de traitement d’ARNt” . Revue annuelle de biochimie . 67 (1): 153–180. doi : 10.1146/annurev.biochem.67.1.153 . PMID 9759486 .
- ^ Ceballos M, Vioque A (2007). “tRNase Z”. Lettres sur les protéines et les peptides . 14 (2): 137–145. doi : 10.2174/092986607779816050 . PMID 17305600 .
- ^ Randau L, Schröder I, Söll D (mai 2008). “La vie sans RNase P”. Nature . 453 (7191): 120–123. Bibcode : 2008Natur.453..120R . doi : 10.1038/nature06833 . PMID 18451863 . S2CID 3103527 .
- ^ Weiner AM (octobre 2004). “Maturation de l’ARNt : polymérisation de l’ARN sans matrice d’acide nucléique” . Biologie actuelle . 14 (20) : R883-5. doi : 10.1016/j.cub.2004.09.069 . PMID 15498478 .
- ^ Kutay U, Lipowsky G, Izaurralde E, Bischoff FR, Schwarzmaier P, Hartmann E, Görlich D (février 1998). “Identification d’un récepteur d’exportation nucléaire spécifique à l’ARNt” . Cellule moléculaire . 1 (3): 359–369. doi : 10.1016/S1097-2765(00)80036-2 . PMID 9660920 .
- ^ Arts GJ, Fornerod M, Mattaj IW (mars 1998). “Identification d’un récepteur d’exportation nucléaire pour l’ARNt” . Biologie actuelle . 8 (6): 305–314. doi : 10.1016/S0960-9822(98)70130-7 . PMID 9512417 . S2CID 17803674 .
- ^ Arts GJ, Kuersten S, Romby P, Ehresmann B, Mattaj IW (décembre 1998). « Le rôle de l’exportine-t dans l’exportation nucléaire sélective des ARNt matures » . La revue EMBO . 17 (24): 7430–7441. doi : 10.1093/emboj/17.24.7430 . PMC 1171087 . PMID 9857198 .
- ^ Yoshihisa T, Yunoki-Esaki K, Ohshima C, Tanaka N, Endo T (août 2003). “Possibilité d’épissage pré-ARNt cytoplasmique : l’endonucléase d’épissage d’ARNt de levure se localise principalement sur les Mitochondries” . Biologie Moléculaire de la Cellule . 14 (8): 3266–3279. doi : 10.1091/mbc.E02-11-0757 . PMC 181566 . PMID 12925762 .
- ^ Kuhnlein, Alexandra; Lanzmich, Simon A.; Brun, Dieter (2 mars 2021). “Les séquences d’ARNt peuvent s’assembler en un réplicateur” . eLife . 10 . doi : 10.7554/eLife.63431 . PMC 7924937 . PMID 33648631 .
- ^ Maximilien, Ludwig (3 avril 2021). “Résoudre le problème de la poule et de l’œuf -” Un pas de plus vers la reconstruction de l’origine de la vie ” ” . SciTechDaily . Récupéré le 3 avril 2021 .
- ^ Kresge, Nicole; Simoni, Robert D.; Colline, Robert L. (7 octobre 2005). “La découverte de l’ARNt par Paul C. Zamecnik” . Journal de chimie biologique . 280 (40) : e37–e39. doi : 10.1016/S0021-9258(20)79029-0 – via www.jbc.org.
- ^ Clark BF (octobre 2006). “La structure cristalline de l’ARNt” (PDF) . Journal des biosciences . 31 (4): 453–457. doi : 10.1007/BF02705184 . PMID 17206065 . S2CID 19558731 .
- ^ Holley RW, Apgar J, Everett GA, Madison JT, Marquisee M, Merrill SH, Penswick JR, Zamir A (mars 1965). “Structure d’un acide ribonucléique”. Sciences . 147 (3664): 1462–1465. Bib code : 1965Sci …147.1462H . doi : 10.1126/science.147.3664.1462 . PMID 14263761 . S2CID 40989800 .
- ^ “Nécrologie” . Le New York Times . 4 juillet 1991.
- ^ “Le Prix Nobel de Physiologie ou de Médecine 1968 : Robert W. Holley – Faits” . Prix Nobel Outreach AB. 2022 . Récupéré le 18 mars 2022 .
- ^ Ladner JE, Jack A, Robertus JD, Brown RS, Rhodes D, Clark BF, Klug A (novembre 1975). “Structure de l’ARN de transfert de phénylalanine de levure à une résolution de 2,5 A” . Actes de l’Académie nationale des sciences des États-Unis d’Amérique . 72 (11): 4414–4418. Bibcode : 1975PNAS…72.4414L . doi : 10.1073/pnas.72.11.4414 . PMC 388732 . PMID 1105583 .
- ^ Kim SH, Quigley GJ, Suddath FL, McPherson A, Sneden D, Kim JJ, Weinzierl J, Rich A (janvier 1973). “Structure tridimensionnelle de l’ARN de transfert de phénylalanine de levure: repliement de la chaîne polynucléotidique”. Sciences . 179 (4070): 285–288. Bibcode : 1973Sci…179..285K . doi : 10.1126/science.179.4070.285 . PMID 4566654 . S2CID 28916938 .
Liens externes
| Wikimedia Commons a des médias liés à TRNA . |
- tRNAdb (version mise à jour et complètement restructurée de la compilation Spritzls tRNA)
- Lien ARNt avec les maladies cardiaques et les accidents vasculaires cérébraux
- GtRNAdb : Collection d’ARNt identifiés à partir de génomes complets
- HGNC : Nomenclature génique des ARNt humains
- Molécule du mois © RCSB Protein Data Bank :
- ARN de transfert
- Aminoacyl-ARNt synthétases
- Facteurs d’allongement
- Entrée Rfam pour l’ARNt